|
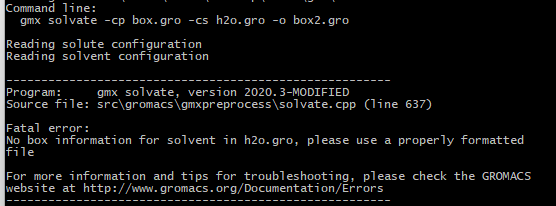
gromacs教程-20-常见建模软件-solvate体系溶剂化 gmx solvate [-cp [<.gro/.g96/..>]] [-cs [<.gro/.g96/…>]] [-o [<.gro/.g96/…>]] [-p [<.top>]] [-nice ] [-box ] [-radius ] [-scale ] [-shell ] [-maxsol ] [-[no]vel] -cp是目的的盒子 -cs需要插入的结构 -o是准备输出结构的名称 gmx editconf -f 2m05.pdb -box 5 5 5 -o box.gro gmx solvate -cp box.gro -cs spc216.gro -o box1.gro -cs 是插入系统自带的水结构,并且会进行充满 gmx solvate -cp box.gro -cs h2o.gro -o box2.gro
当系统的文件没有时则无法添加 gmx solvate -cp box.gro -cs tip4p.gro -o box2.gro gmx solvate -cp box.gro -cs tip5p.gro -o box3.gro
系统的文件中含有的结构
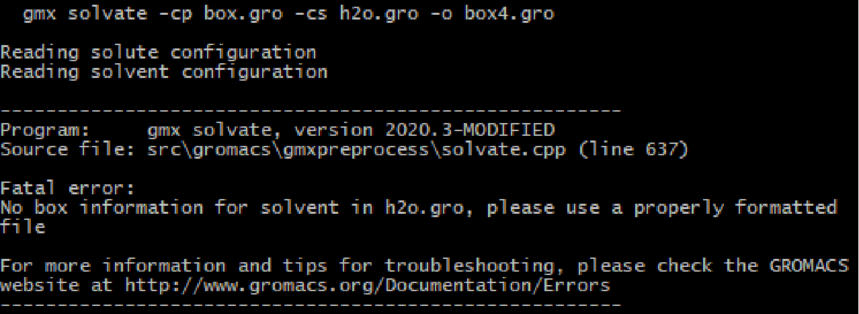
gmx solvate -cp box.gro -cs h2o.gro -o box4.gro
把要添加到的结构添加到系统文件夹中 依然发现好像还是没有啥用处 查看solvate.cpp 637行,暂时还没看懂怎么修改程序 先暂时放在这,大家需要插入h2o等没有的分子 可以用gmx insert-molecules
gmx solvate -cp box.gro -cs spc216.gro -o box4.gro -maxsol 200 -maxsol 加入的最大溶剂分子数. 若为零(默认)则忽略此选项,设置200则只插入了200分子,比之前确实少很多,这个和gmx insert-molecules中的-nmol 很像
gmx solvate -cp box.gro -cs spc216.gro -o box5.gro -shell 5 gmx solvate -cp box.gro -cs spc216.gro -o box6.gro -shell 1 -shell 溶质周围水层的可选厚度,单位是nm,已知盒子大小3nm,现在设置5nm水层明显超过了盒子大小,那么它只会充满盒子,成为盒子形状,不会超出盒子,如果体系+水层低于3nm,则会像第2个图一样呈现出体系的形状
gmx solvate -cp box.gro -cs spc216.gro -o box7.gro -maxsol 100 -radius 10000 gmx solvate -cp box.gro -cs spc216.gro -o box8.gro -maxsol 100 -radius 1 -radius 默认范德华距离0.105nm,设置完其实没看出太大的影响,反正建议大家在不清楚前不进行设置,gmx insert-molecules也是这样的噢,不清楚就不要设置
gmx solvate -cp box.gro -cs spc216.gro -o box9.gro -scale 1 gmx solvate -cp box.gro -cs spc216.gro -o box10.gro -scale 0.1 -scale默认值0.57,变大变小会对插入的物质进行发生变化,如果输入0.1变小,那么水与里面的蛋白会发生位置识别错误,因此大家使用时注意一下
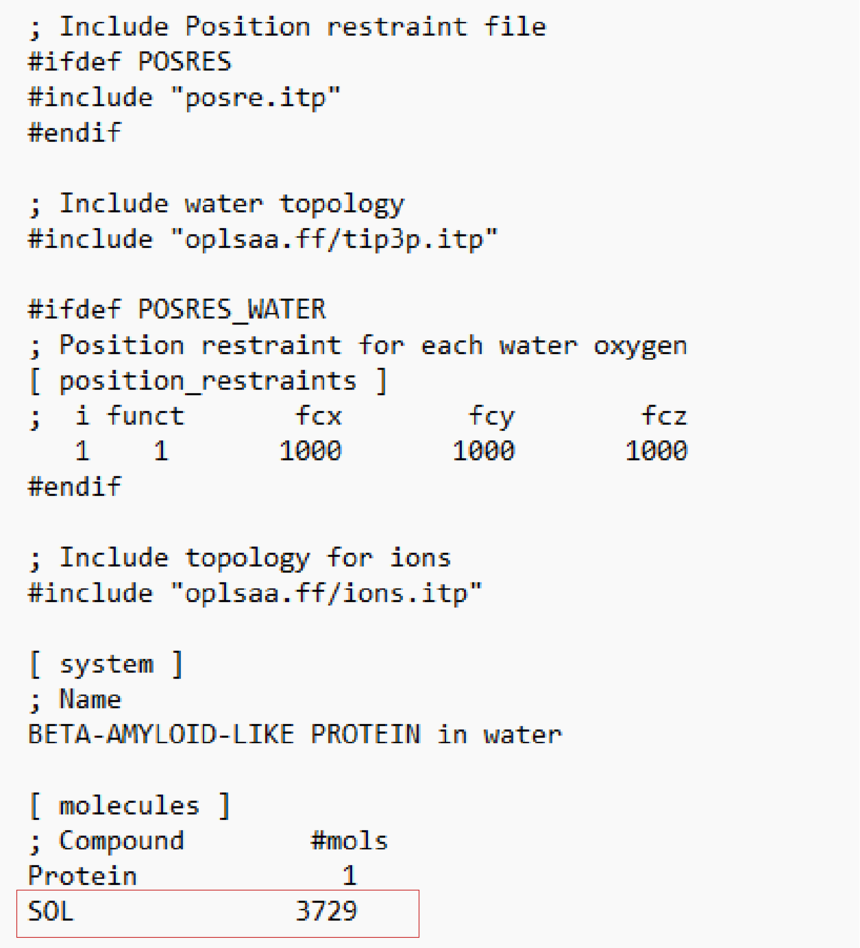
gmx pdb2gmx -f box.gro -ignh gmx solvate -cp conf.gro -cs spc216.gro -o box11.gro -p topol.top -p更新top文件,建议大家在使用过程中最好使用,免得自己手动去加水
-nice表示设置优先级 -[no]vel保持溶质和溶剂分子输入文件中的速度,这个选项注意一下以后可能用的上,编写时暂时不知道咋用,但是感觉还是很有用处的,因为MD中速度时很关键的 |