|
gromacs教程-25-常见建模软件-packmolPackmol的官网https://m3g.github.io/packmol/ Packmol的书写格式如下 tolerance 2.0 表示原子间的距离不能小于2.0A add_box_sides 1.2 表示给盒子的方向增加1.2A,目的是为了避免边界的原子和镜像盒子里的原子间存在不合理接触 output xx1.pdb 表示输出xx1.pdb文件, structure xx2.pdb 表示输入xx2.pdb结构 number xxx 表示输入的xx2.pdb一共xxx个数目 inside box x y z 表示输入盒子的尺寸,可用的盒子cube/box/sphere/cylinder,其中inside与outside可换 还可使用over(或below) plane/constrain_rotation/fixed end structure 结束xx2.pdb输入结束 structure xx3.pdb number xxx atoms xxx 原子规则,原子xxx号服从下面的规则 inside box x y z end atoms 原子规则结束 end structure *filetype xyz 表示可以输出文件类型xyz *seed -1 表示每次运行产生的结构都会不同 约束规则 inside cube [xmin ymin zmin d]:要求分子出现在长度为d的立方盒子内,盒子x,y,z最小值为xmin ymin zmin inside box [xmin ymin zmin xmax ymax zmax]:要求分子出现在矩形盒子内,盒子两个顶角坐标分别为xmin ymin zmin和xmax ymax zmax inside sphere [x y z r]:要求分子出现在中心为(x,y,z)半径为r的圆球中。另外,用inside ellipsoid关键词的话还可以要求出现在特定的椭球中 inside cylinder [a1 b1 c1 a2 b2 c2 r l]:要求分子出现在圆柱内。圆柱的定义是从(a1,b1,c1)往(a2,b2,c2)方向伸展l长度,半径为r inside ellipsoid a1 b1 c1 a2 b2 c2 d:坐标(a1,b1,c1)定义椭球面的中心, 坐标(a2,b2,c2)定义坐标轴的相对大小, d定义椭球的大小. 以上inside都可以改为outside,要求不能出现在指定范围内。上面的空间范围要求可以同时使用多个,条件会同时满足。还有一些设置: over plane [a b c d]:要求分子出现位置满足平面方程ax+by+cz-d>=0。比如over plane 1 0 0 10.5就相当于要求出现在x>=10.5埃的区域。over可以改成below来反转条件。 constrain_rotation [x/y/z 平均值 最大偏差]:默认情况下分子可以绕x,y,z轴随意旋转任意角度。用这个选项可以设置分子绕x或y或z旋转的情况,可以同时设多个。如constrain_rotation x 20 5代表允许绕着y和z轴随意旋转,而绕着x轴旋转的范围必须在15~25度之间,平均值为20度。 fixed [x y z a b c]:用来直接定义分子出现在哪里。此设置代表将分子结构文件里的坐标平移(x,y,z),并绕x,y,z轴分布旋转a,b,c弧度。如果还同时写了center关键词,相当于把分子的中心摆到(x,y,z)位置再旋转 *packmol < xxx.inp运行输入 用cmd运行,powershell和gitbash没有数据库无法使用 tolerance 10.0 add_box_sides 1.2 output toler.pdb structure s4.pdb number 50 inside cube 0. 0. 0. 50. end structure
tolerance 2.0 add_box_sides 5 output sides.pdb structure s4.pdb number 50 inside cube 10. 10. 10. 50. end structure
*add_box_sides不是非得加,大部分时候是可以不需要进行添加的。 tolerance 2.0 add_box_sides 1.2 output cube1.pdb structure s4.pdb number 50 inside cube 0. 0. 0. 50. end structure tolerance 2.0 add_box_sides 1.2 output cube3.pdb structure s4.pdb number 50 inside cube 10. 10. 10. 50. end structure tolerance 2.0 add_box_sides 1.2 output cube2.pdb structure s4.pdb number 100 inside cube 0. 0. 0. 50. end structure
tolerance 2.0 add_box_sides 1.2 output ellipsoid.pdb structure h2o.pdb number 4000 inside ellipsoid 0. 0. 0. 20. 20. 20. 50. end structure
tolerance 2.0 add_box_sides 1.2 output ellipsoid2.pdb structure h2o.pdb number 4000 inside ellipsoid 0. 0. 0. 1. 2. 3. 10. end structure
tolerance 2.0 add_box_sides 1.2 output box1.pdb structure h2o.pdb number 2000 inside box 0. 0. 0. 30. 40. 50. end structure tolerance 2.0 add_box_sides 1.2 output box3.pdb structure h2o.pdb number 2000 inside box 10. 10. 10. 40. 50. 60. end structure tolerance 2.0 add_box_sides 1.2 output box2.pdb structure h2o.pdb number 2000 inside box 10. 10. 10. 30. 40. 50. end structure
tolerance 2.0 add_box_sides 1.2 output sphere.pdb structure h2o.pdb number 3000 inside sphere 0. 0. 0. 50. end structure
tolerance 2.0 add_box_sides 1.2 output cylinder.pdb structure h2o.pdb number 5000 inside cylinder 0. 0. 0. 50. 50. 50. 25. 50. end structure
tolerance 2.0 add_box_sides 1.2 output o-sphere.pdb structure s4.pdb number 50 outside sphere 0. 0. 0. 50. end structure tolerance 2.0 add_box_sides 1.2 output o-cube.pdb structure s4.pdb number 50 outside cube 0. 0. 0. 50. end structure tolerance 2.0 add_box_sides 1.2 output o-cylinder.pdb structure s4.pdb number 50 outside cylinder 0. 0. 0. 50. 50. 50. 25. 5. end structure 在没有做出具体的盒子大小前直接使用outside是不科学的 大家可以吃饱了没啥事进行试试看。 tolerance 2.0 add_box_sides 1.2 output o-plane1.pdb structure h2o.pdb number 2000 inside cube 0. 0. 0. 50. over plane 1 0 0 10 end structure tolerance 2.0 add_box_sides 1.2 output b-plane.pdb structure h2o.pdb number 2000 inside cube 0. 0. 0. 50. below plane 1 0 0 10 end structure tolerance 2.0 add_box_sides 1.2 output o-plane2.pdb structure h2o.pdb number 2000 inside cube 0. 0. 0. 50. over plane 5 5 5 10 end structure tolerance 2.0 add_box_sides 1.2 output o-plane3.pdb structure h2o.pdb number 2000 over plane 1 0 0 10 end structure *没有设定盒子前这个over也是没有用的
tolerance 2.0 add_box_sides 1.2 output cube-cube1.pdb structure s4.pdb number 50 inside cube 0. 0. 0. 50. end structure structure h2o.pdb number 1000 inside cube 50. 50. 50. 50. end structure
tolerance 2.0 add_box_sides 1.2 output cube-cube2.pdb structure s4.pdb number 50 inside cube 0. 0. 0. 50. end structure structure h2o.pdb number 1000 inside cube 0. 50. 50. 50. end structure
tolerance 2.0 add_box_sides 1.2 output cube-cube3.pdb structure s4.pdb number 50 inside cube 0. 0. 0. 50. end structure structure h2o.pdb number 1000 inside cube 0. 0. 0. 50. end structure
tolerance 2.0 add_box_sides 1.2 output sphere-sphere1.pdb structure s4.pdb number 50 inside sphere 0. 0. 0. 50. end structure structure h2o.pdb number 1000 inside sphere 50. 50. 50. 50. end structure tolerance 2.0 add_box_sides 1.2 output sphere-sphere2.pdb structure s4.pdb number 50 inside sphere 0. 0. 0. 50. end structure structure h2o.pdb number 1000 inside sphere 20. 20. 20. 50. end structure tolerance 2.0 add_box_sides 1.2 output sphere-sphere3.pdb structure s4.pdb number 50 inside sphere 0. 0. 0. 50. end structure structure h2o.pdb number 3000 inside sphere 0. 0. 0. 70. end structure tolerance 2.0 add_box_sides 1.2 output cube-cube22.pdb structure h2o.pdb number 5000 inside cube 0. 0. 0. 70. outside cube 0. 0. 0. 40. end structure
tolerance 2.0 add_box_sides 1.2 output cube-cube23.pdb structure h2o.pdb number 5000 inside cube 0. 0. 0. 70. outside sphere 0. 0. 0. 20. end structure
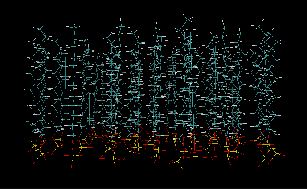
tolerance 2.0 output mem1.pdb structure SDS.pdb number 64 inside cube 0. 0. 0. 40. atoms 39 over plane 0. 0. 1. 16. end atoms atoms 1 below plane 0. 0. 1. 32.4 end atoms end structure
tolerance 2.0 output mem2.pdb structure SDS.pdb number 64 inside cube 0. 0. 0. 40. atoms 39 below plane 0. 0. 1. 16. end atoms atoms 1 over plane 0. 0. 1. 32.4 end atoms end structure
tolerance 2.0 output mem3.pdb structure SDS.pdb number 4 inside cube 0. 0. 0. 40. atoms 39 below plane 0. 0. 1. 16. end atoms atoms 1 over plane 0. 0. 1. 32.4 end atoms end structure
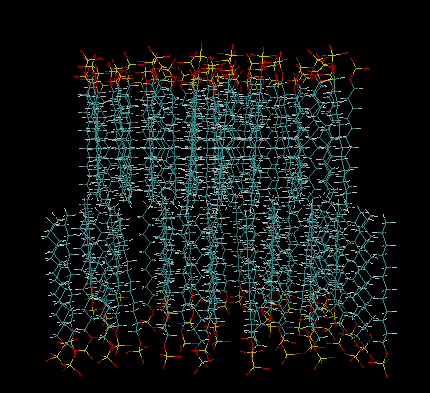
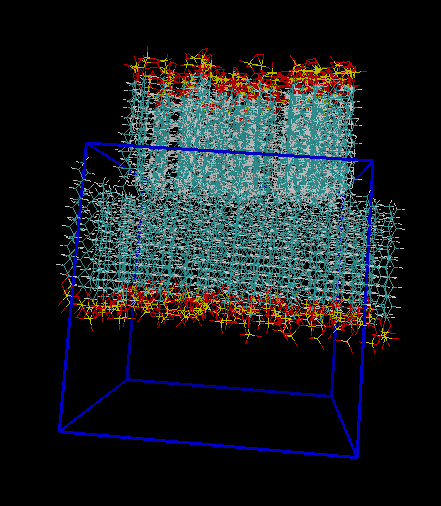
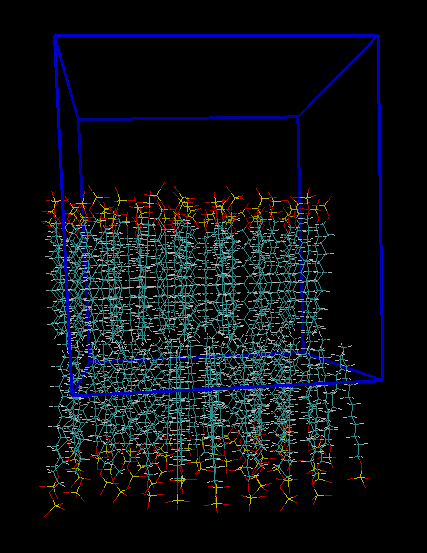
tolerance 2.0 output mem4.pdb structure SDS.pdb number 60 inside box 0. 0. 0. 40 40 80. atoms 39 below plane 0. 0. 1. 16. end atoms atoms 1 over plane 0. 0. 1. 32.4 end atoms end structure structure SDS.pdb number 60 inside box 0. 0. 0. 40 40 80. atoms 39 over plane 0. 0. 1. 50.8 end atoms atoms 1 below plane 0. 0. 1. 34.4 end atoms end structure
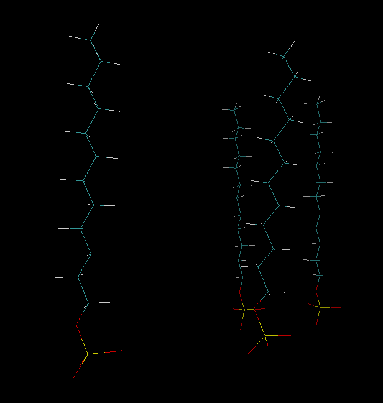
atoms 39/1分别是主链上的头和尾 vmd测量距离约为16.4A tolerance 2.0 output mem5.pdb add_box_sides structure SDS.pdb number 200 inside box 0. 0. 0. 40 40 80. atoms 39 below plane 0. 0. 1. 16. end atoms atoms 1 over plane 0. 0. 1. 32.4 end atoms end structure structure SDS.pdb number 200 inside box 0. 0. 0. 40 40 80. atoms 39 over plane 0. 0. 1. 50.8 end atoms atoms 1 below plane 0. 0. 1. 34.4 end atoms end structure
tolerance 2.0 output mem6.pdb add_box_sides structure SDS.pdb number 64 inside box 0. 0. 0. 40. 40. 80. atoms 39 below plane 0. 0. 1. 16. end atoms atoms 1 over plane 0. 0. 1. 32.4 end atoms end structure structure SDS.pdb number 64 inside box 0. 0. 33.4. 40. 40. 80. atoms 39 over plane 0. 0. 1 50.8 end atoms atoms 1 below plane 0. 0. 1 34.4 end atoms end structure
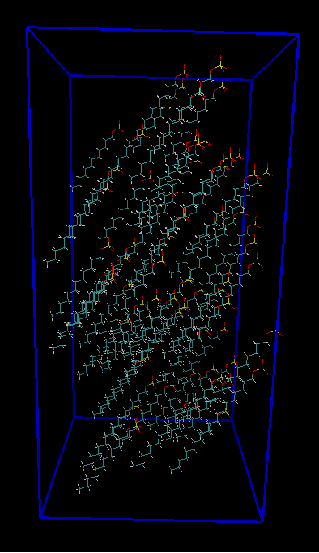
tolerance 2.0 output mem7.pdb add_box_sides structure SDS.pdb number 64 inside box 0. 0. 0. 40. 40. 80. constrain_rotation x 90. 0. constrain_rotation y 90. 0. constrain_rotation z 90. 0. end structure
tolerance 2.0 output qiu.pdb structure h2o.pdb number 1200 inside sphere 0. 0. 0. 20. end structure structure SDS.pdb number 200 atoms 39 inside sphere 0. 0. 0. 22. end atoms atoms 1 outside sphere 0. 0. 0. 38.4 end atoms end structure
resnumbers 0 这种情况下, 所有残基编号会对应于每类分子, 与原始pdb文件中的残基编号无关. 这意味着, 如果你堆积10个水分子和10个乙醇分子, 水分子的编号为1到10, 乙醇分子的编号也是1到10. resnumbers 1 这种情况下, 会保持原始pdb文件中的残基编号不变. 这意味着如果你堆积10个含有5个残基的蛋白质, 残基编号会被保留, 因此, 在相同蛋白质的每个分子中, 等价残基的编号会重复. resnumbers 2 在这种情况下, 结构中所有残基的残基编号会根据相同文件中前面列出残基编号按顺序编号. 这意味着, 这意味着如果你堆积10个含有5个残基的蛋白质, 残基编号会从1到50. resnumbers 3 在这种情况下, 残基编号相应于文件中所有残基的序列号. 也就是, 如果你堆积一个含有150个残基的蛋白质, 外加10个水分子, 水分子的编号为从151到161. discale [实数] 此选项控制局部优化方法中实际使用的距离容差. 我们发现使用大的距离有时会有帮助. 例如, 试着将discale设为1.5. maxit [整数] 这是每次循环中局部优化(GENCAN)的最大迭代次数. 目前的默认值是20, 改变此值收敛情况可能更好(或更差) movebadrandom Packmol的一种收敛策略是移动那些放置得不好的分子, 如果设定了这个选项, 分子将会被放置在盒子中新的随机位置上. 否则(默认)分子会被移动到堆积好的分子的附近位置. 当限制很复杂时, 使用此选项可能会有帮助, 但当结构很大时, 可能很差, 因为新的随机位置可能与那些重叠. * https://wap.sciencenet.cn/blog-548663-994817.html?mobile=1在分子之间添加TER标识(AMBER使用此选项): add_amber_ter 增加盒子边长信息到输出的PDB文件中(GROMACS使用此选项): add_box_sides 1.0. 其中1.0为可选的实数, 为每边增加的长度. 如果模拟盒子实际的边并不精确地等于体系中分子的最大最小坐标(通常, 如果你使用周期性边界条件, 你可能想要增加1.0来避免边界处的碎裂). 增加最大体系的尺寸: sidemax [实数] (如: sidemax 2000.d0) sidemax用于构建分子分布的初始近似. 如果你的体系非常大, 或者看起来被最大尺度切断了, 请增加sidemax. 体系的所有坐标必须处于-sidemax和+sidemax之间. 使用大致符合体系的sidemax, 可能会加速堆积计算. 默认值为1000.d0. 改变随机数产生器的种子: seed [整数] (如: seed 191917) 使用-1会根据计算机时间自动生成种子 使用真正随机的初始点来最小化(默认选项是生成一个均匀密度的初始点, 固定分子之间没有重叠):randominitialpoint 改变每个循环中Gencan迭代的最大数目: maxit [整数] 改变循环的最大数目: nloop [整数] 改变输出文件写入的频率: writeout [整数] 将当前结构写入输出文件, 即便它比目前的最佳结构更差(仅用于检查目的): writebad 检查初始点, 仅用于测试目的, 仅构建初始近似, 写入输出文件, 然后退出: check 修改优化例程打印输出: iprint1 [整数] 和/或 iprint2 [整数] 整数必须是0 1 2 3 修改连接分胞方法的分格数目(技术): fbins [实数] 默认值为根号3 比较解析和有限差分的梯度. 仅仅用于测试的目的. 将比较结果写入chkgrad.log文件: chkgrad *https://wap.sciencenet.cn/blog-548663-994817.html?mobile=1 |