|
gromacs教程-18-常见建模软件-gmx editconfgmx editconf [-f [<.gro/.g96/...>]] [-n [<.ndx>]] [-o [<.gro/.g96/...>]] [-mead [<.pqr>]] [-bf [<.dat>]] [-nice ] [-[no]w] [-[no]ndef] [-bt ] [-box ] [-angles ] [-d ] [-[no]c] [-center ] [-aligncenter ] [-align ] [-translate ] [-rotate ] [-[no]princ] [-scale ] [-density ] [-[no]pbc] [-resnr ] [-[no]grasp] [-rvdw ] [-[no]sig56] [-[no]vdwread] [-[no]atom] [-[no]legend] [-label ] [-[no]conect] 这个命令是用来调整坐标,设定盒子形状以及尺寸大小的,在做分子动力学时,一定时要加盒子的,盒子大小决定了你容纳的物质,盒子的形貌也会影响到计算性能,以及其他的情况,所以盒子最好建的合理,最大不要超过15nm,如果要超过建议你用lammps去考虑,因为建的太大容易麻烦,不知道形状就考虑正方体,当然这个不是一个比较好的方式。 -f选项表示需要处理哪个分子 -o选项表示处理完输出的结构名字 不一定非要用来建盒子,可以通过gmx editconf进行处理分子的坐标,这样可以用于和脚本配合进行生成你需要的模型 gmx editconf -f 2m05.pdb -o box1.gro -box 8 6 5 -box选项表示为自定义盒子尺寸大小
gmx editconf -f 2m05.pdb -o box2.gro -box 8 6 5 -rotate 0 0 90 -rotate选项表示输入物质旋转,不是盒子旋转 0 0 90表示垂直x轴旋转0 垂直y轴旋转0 垂直z轴旋转90
gmx editconf -f pfha.pdb -o box3.gro -princ -princ 让分子与x轴尽量平行,但是没那么准,还是会有点歪的,旋转这个分子体系就好了,输入编号,按回车
*看见没有,这个是可以不加入盒子的噢
gmx editconf -f 2m05.pdb -o box4.gro -bt triclinic gmx editconf -f 2m05.pdb -o box5.gro -bt triclinic -box 8 6 5 -bt选项表示盒子形状,但是不涉及到相关的尺寸与大小,所以上述两种情况做出来的话都有问题 triclinic跟着-bt后面表示盒子为三斜盒子
gmx editconf -f 2m05.pdb -o box6.gro -bt triclinic -box 8 6 5 -angles 45 60 90 设置了-angles和-box才能提现出盒子的三斜形式,当然上述的参数纯粹扯淡,因为你没法成为周期性,所以这个参数需要你们自己衡量形成一个周期性的数值 -angles表示盒子的三个角度
gmx editconf -f 2m05.pdb -o box7.gro -bt triclinic -d 8 -angles 45 60 90 -d选项表示指定体系中的原子到盒子编边界的最小距离. 使用-d选项时, 对三斜盒子会使用体系在x, y和z方向的大小, 对立方盒子, 菱形十二面体盒子或截角八面体盒子, 盒子的大小被设定为体系直径(原子间的最大距离)加上两倍的指定距离,记住这个是替代-box,不是完整的替代-box 和 -angles的
gmx editconf -f 2m05.pdb -o box8.gro -bt dodecahedron -box 7 -angles 60 dodecahedron菱形十二面体盒子(等边十二面体) -box 后续只需要加入一个数字就好,因为这个是正的 -box 7 8 9 默认只识别7,也就是第一个数字,-angles 输入多少都没有啥用,因为这个角度是固定的
gmx insert-molecules -f box8.gro -ci h2o.gro -nmol 50000 -o box9.gro
gmx insert-molecules -f box6.gro -ci h2o.gro -nmol 50000 -o box10.gro
gmx editconf -f 2m05.pdb -o box11.gro -bt octahedron -d 4 octahedron为截角八面体盒子(即将两个底面重合的四面体切去方向相反的两头, 同时保证所有的边长相等)截角八面体三个盒向量的长度是两个相对六边形之间的最短距离. 相对于具有周期性映象距离的立方盒子, 具有相同周期距离的菱形十二面体盒子的体积是立方盒子的71%, 而截角八面体盒子的体积是立方盒子的77%.
gmx editconf -f 2m05.pdb -o box11.gro -box 5 5 5 -center 0 0 0 gmx editconf -f 2m05.pdb -o box12.gro -box 5 5 5 -center 0 5 0
gmx editconf -f 2m05.pdb -o box11.gro -box 5 5 5 -nopbc -nopbc与-pbc no是一个意思,去除周期性的子程序可能会崩溃,可以粗略地去除体系的周期性. 当去除周期性时, 输入文件最底部的盒向量必须保证正确, 这非常重要, 因为gmx editconf去除周期性的算法十分简单, 只是将原子坐标直接减去盒子边长. gmx editconf -f 2m05.pdb -o box12.gro -box 5 5 5 -density 2000 缩放会在任何其他操作之前进行. 可以对盒子和坐标进行缩放以得到一定的密度(选项-density). 注意如果输入是.gro文件的话, 密度可能不够精确. gmx editconf -f 2m05.pdb -o box12.pdb -box 5 5 5 -density 10000000 gmx editconf -f 2m05.pdb -o box13.pdb -box 5 5 5 -density 1000000000 通过缩放使输出盒子的密度(g/L=10-3g/cm^3=相对原子质量*0.602 g/nm^3 mol)为指定值 假设单个体系相对原子质量10000g/mol 10000*0.602*5*5*5=752500
gmx editconf -f 2m05.pdb -o box13.gro -box 5 5 5 -scale 10 10 10 gmx editconf -f 2m05.pdb -o box14.gro -box 5 5 5 -scale 1 1 1 gmx editconf -f 2m05.pdb -o box15.gro -box 5 5 5 -scale 1 0.2 0.5 -scale选项的一个特性是, 当某一维度的缩放因子为-1时, 可以得到体系相对于一个平面的镜面映象. 当三个维度的缩放因子都是-1时, 可以获得体系相对于坐标原点的对称映象.没啥太多事不加这个噢,你看1 2 5,相当于分子沿x维持1倍,y变成0.2倍,z变成0.5倍。我暂时还没想到这个-sacle用来干嘛,后期想到了,就会加上去,只能说这个可以让大家适当的调整结构的膨胀收缩。
gmx editconf -f h2o -o box16.gro -box 5 5 5 gmx insert-molecules -f box16.gro -ci h2o.gro -nmol 1000 -o box17.gro gmx editconf -f box17.gro -o box18.gro -density 10000 gmx editconf -f box17.gro -o box19.gro -density 1000 gmx editconf -f box17.gro -o box20.gro -density 100
gmx editconf -f 2m05.pdb -o box21.gro -box 8 6 5 -align 4 0 0 -align选项表示体系与目标向量进行平齐
gmx editconf -f 2m05.pdb -o box22.gro -box 8 6 5 -rotate 90 0 0 gmx editconf -f 2m05.pdb -o box23.gro -box 8 6 5 -aligncenter 4 4 4 -rotate 90 0 0 gmx editconf -f 2m05.pdb -o box24.gro -box 8 6 5 -aligncenter 4 0 0 -rotate 90 0 0 选项-align可以将特定组的主轴与给定的向量平齐, -aligncenter选项指定可选的旋转中心.
gmx editconf -f 2m05.pdb -o box25.gro -rotate 90 0 0 gmx editconf -f 2m05.pdb -o box26.gro -aligncenter 4 4 4 -rotate 90 0 0 gmx editconf -f 2m05.pdb -o box27.gro -aligncenter 4 0 0 -rotate 90 0 0 -aligncenter选项表示平齐的旋转中心,上述输入完全重合
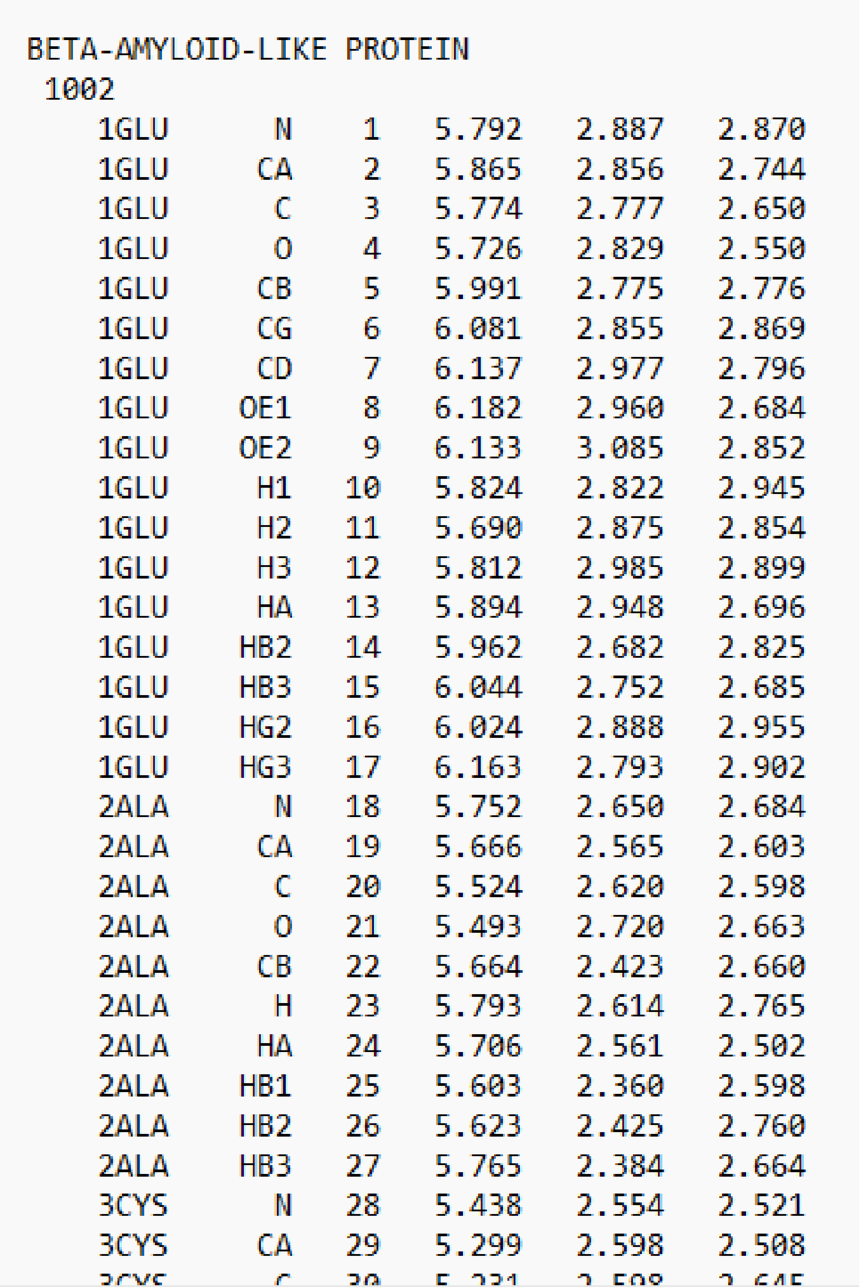
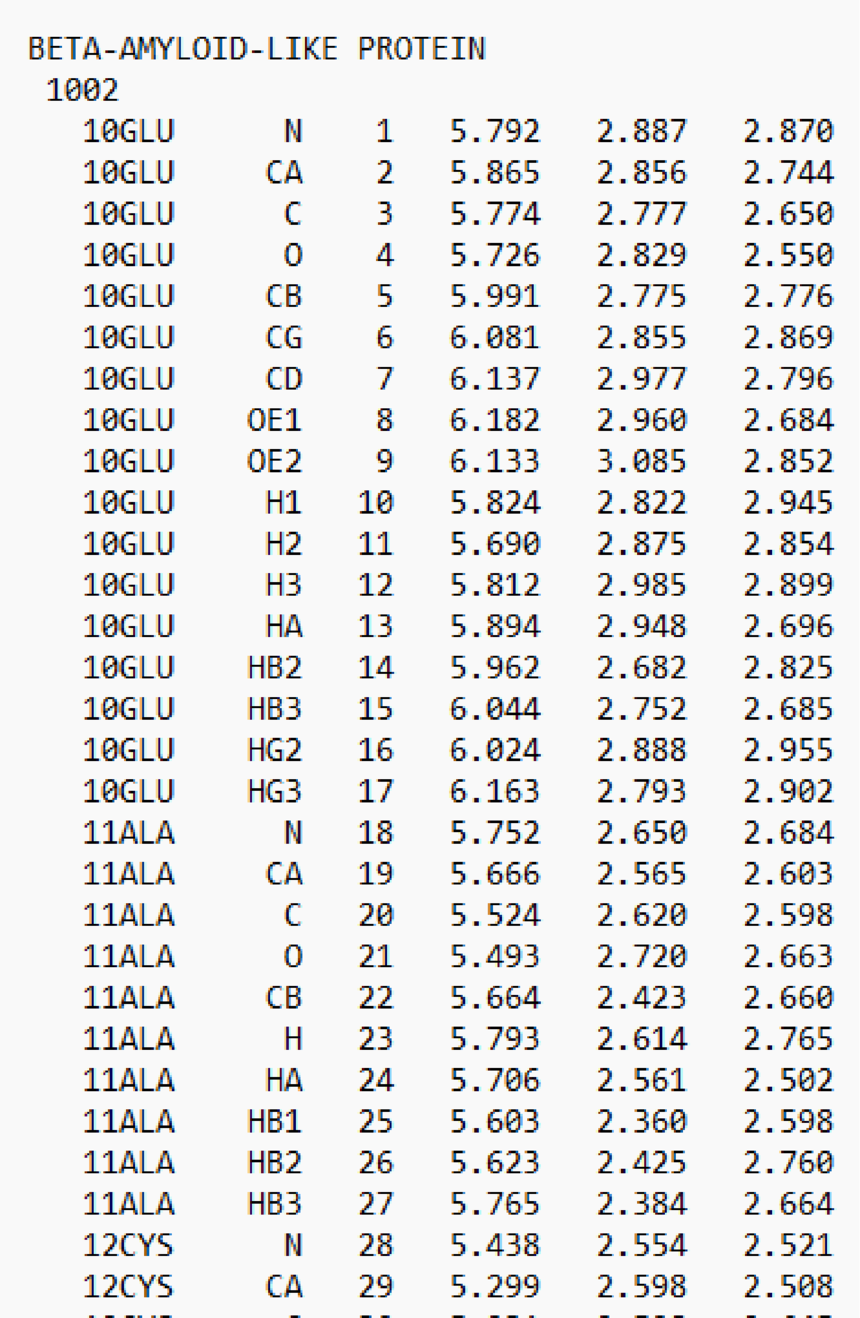
gmx editconf -f box1.gro -o box28.gro -resnr 1 从resnr开始重新对残基进行编号 gmx editconf -f box1.gro -o box29.gro -resnr 10
gmx make_ndx -f 2m05.pdb -o index.ndx 当使用-n时, 可以指定一个索引文件, 并选择其中的一个组来计算大小和几何中心, 否则会使用整个体系的大小和几何中心.或-ndef时组的选择是在其他所有操作都完成之后进行的. 在程序输出时, 可以只输出体系中的某一个组, 或者某一个部分, 还可以建立划分更细致的索引文件, 以便进行更加细致的选择. -princ这个选项可以用来对齐分子, 比如使分子沿X轴对齐. 例如, 你想将分子中的两个残基沿Y轴对齐, 那么就在索引文件中将这俩个残基标记一下, 然后使用-princ, 根据提示就能对齐分子了. 当输出.pdb文件时, 可以使用-bf选项添加B因子. B因子可以从文件中读取, 格式如下: 第一行声明文件中所含B因子数值的个数, 从第二行开始, 每行声明一个索引号, 后面跟着B因子. 默认情况下, B因子将附加到每个残基上, 每个残基一个数值, 除非索引大于残基数目或者设定了-atom选项. 显然, 可以添加任何类型的数值数据而不仅仅是B因子. -legend选项将生成一列CA原子, 其B因子的范围为所用数据的最小值到最大值, 可以有效地作为查看的图例, 便于可视化软件显示. 使用-mead选项时可以生成一个特殊的.pdb文件(.pqr), 它可用于MEAD静电程序(泊松玻尔兹曼方程求解器). 使用这个选项的前提条件是输入文件必须为运行输入文件(如tpr), 因为这样的文件中才包含了力场参数. 输出文件中的B因子段为原子的范德华半径而占有率段为原子的电荷. -grasp选项的作用与上一选项类似, 只不过互换了电荷与半径的位置, 电荷位于B因子段, 而半径位于占有率段. -conect选项当写入的时候将CONECT记录添加到.pdb文件中. 只有当拓扑文件存在时才可以 -rvdw选项如果在数据库中找不到范德华半径或者拓扑文件中不存在参数, 将使用默认的范德华半径(单位nm).可用于处理缺少力场参数的原子. -novdwread选项从vdwradii.dat文件中读取范德华半径, 而不是根据力场计算半径. 最后, 使用选项-label, gmx editconf可以为.pdb文件添加一个链标识符. 如果一个文件中不同残基属于不同肽链, 那么这个选项可以为残基指定肽链归属, 这样不但有利于可视化, 在使用一些程序如Rasmol进行分析时也很有帮助, 在建立模拟体系时也十分方便. |