|
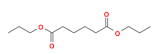
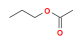
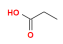
LAMMPS讲解71-ReaxFF力场验证ReaxFF已经存在很多元素的力场参数。很多时候能够找到包含所需元素的力场参数。但是力场参数往往是根据特定体系而开发的,是否适用于自己的体系则需要验证力场参数。验证力场参数的基本流程就是对比自己体系中的分子各种能量(如键,键角,二面角)与DFT计算的能量。如果吻合较好那么找到的力场参数就可以用,如果不好就要基于找到的力场参数进行进一步优化。本节介绍力场参数的验证过程。严格的验证力场需要对比各项能量和参数,包括键结、非键结和电荷平衡参数。 (1)电荷平衡参数验证 这个可以通过计算一个完整分子和分子片段内原子的电荷,对比DFT和ReaxFF的计算结果。以合成酯为例,合成酯中只包含CHO三种元素。可以使用这篇文章(Chowdhury Ashraf and Adri C.T. van Duin* Extension of the ReaxFF Combustion Force Field toward Syngas Combustion and Initial Oxidation Kinetics J. Phys. Chem. A 121, 5, 1051-1068)中所开发出的力场参数。把力场参数下载下来,命名为ffield2016.reax.cho。目前在LAMMPS中只用Qeq方法进行电荷动态平衡。该方法要求有三个参数分别是 chi eta gamma 对于LAMMPS来说,可以直接采用ReaxFF中力场文件中的参数,也可以自己单独写一个文件定义这些参数。通常我们会直接采用力场文件中的参数。这里要验证的就是力场文件中的这三个参数。对于合成酯采用以下几个分子进行验证。
分子1 分子2 分子3 为什么要选择这几种。这就需要对研究对象有充分的了解。这里要研究合成酯的热解问题,那么首先合成酯的完整分子自然要考虑,一半合成酯也可以考虑一下。然后根据前人研究合成酯会发生以下热解过程,那么热解过程中的反应产物也要考虑。而不含O的自由基和分子就不用考虑了,因为待验证的力场在开发时已经充分验证了。
这里采用LAMMPS计算ReaxFF所得到的电荷并与采用量子化学的电荷进行对比。要计算首先要有输入文件的坐标。可以采用Avogadro进行坐标生成。这里没有采用MS是因为MS太大了,这么个小事不需要这么大的软件。而且MS收费,使用收费软件不符合讲义特点。使用Avogadro分别生成上述三个分子的pdb文件和xyz文件。在Avogadro中画完分子之后一定要做几下结构优化只有再导出文件。 Qeq方法只需要分子几何构型和原子的实验性质就可以预测分子内部的每个原子的电荷。先将导出的pdb文件中转换为data文件(一个分子就够了),然后在lammps中计算每个原子的部分电荷。计算in文件如下 units real atom_style full neighbor 2.0 bin neigh_modify every 1 delay 0 check no read_data half.data pair_style reaxff lmp_control pair_coeff * * ffield2016.reax.cho C H O timestep 0.25 velocity all create 300.0 4928459 rot yes dist gaussian thermo 100 thermo_style custom step temp pe ke thermo_modify flush yes fix 1 all nve fix 2 all temp/berendsen 300.0 300.0 50.0 fix 3 all qeq/reaxff 1 0.0 10.0 1e-6 param.qeq run 100000 dump 1 all custom 100 dump.lammpstrj id mol type q x y z dump_modify 1 sort id run 100000 计算完成后将dump中的电荷取时间平均,对应的python脚本为 input_file = open('E:\\work\\ester_reax\\diester\\力场验证\\third\\dump.lammpstrj','r') output_file = open('E:\\work\\ester_reax\\diester\\力场验证\\third\\aveq','w') natom = 11 aveq = [[0]*2 for _ in range(natom)] line = input_file.readline() tmp = "" no = 0 while line: if line == "ITEM: TIMESTEP\n": line = input_file.readline() line = input_file.readline() line = input_file.readline() line = input_file.readline() line = input_file.readline() line = input_file.readline() line = input_file.readline() line = input_file.readline() for i in range(natom): line = input_file.readline().split() aveq[i][0] += float(line[3]) no += 1 line = input_file.readline()
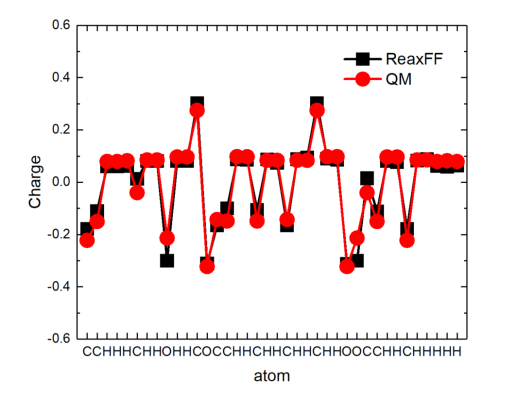
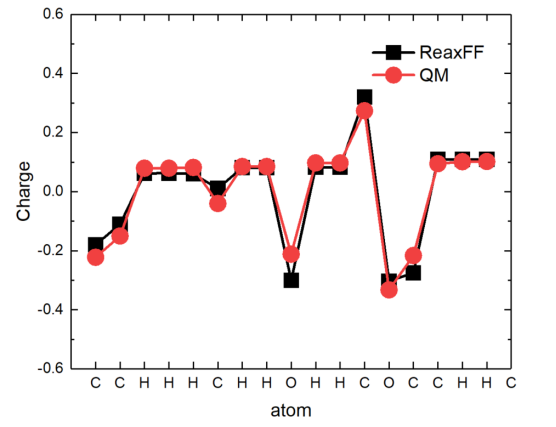
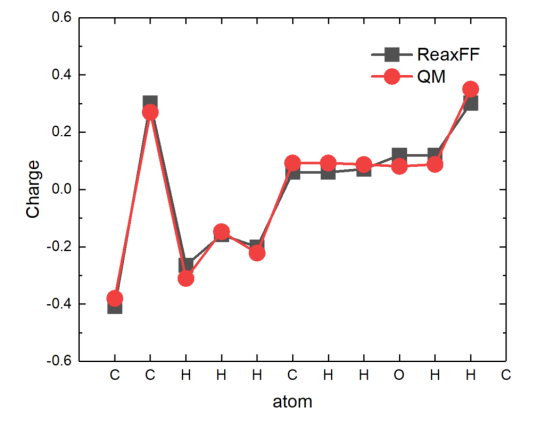
for i in range(natom): aveq[i][0] = aveq[i][0]/no output_file.write(str(aveq[i][0]) + "\n") output_file.close() 这样就能得到有ReaxFF计算得到的电荷。然后使用导出的xyz文件,调用Multiwfn软件和Gaussian程序使用量子化学计算得到的电荷。这里我们采用1.2CM5方法计算电荷。计算的采用的量子化学信息为B3LYP-D3(BJ)/def2-SVP。由于1.2*CM5电荷就是在Truhlar等人提出的原始的CM5电荷基础上乘上1.2,这相当于增大了原子电荷的数量级,等效地体现出了溶剂环境对溶质的极化作用。这里的模拟没有溶剂化作用,所以需要对得到的电荷除以1.2得到CM5电荷。关于如何联用Multiwfn软件和Gaussian程序计算CM5电荷,参见网址计算适用于OPLS-AA力场做模拟的1.2*CM5原子电荷的懒人脚本 - 思想家公社的门口:量子化学·分子模拟·二次元 (sobereva.com)。这里要求Gaussian程序是Linux版下的。如需获取Linux版的Gaussian可加微信baolu_yao。 以下是上述三个分子中ReaxFF计算得到的原子电荷和量子化学得到的原子电荷对比。从三个图中可以看到这里所采用的Qeq参数以很高的精度再现了量子化学计算得到的电荷,表明采用Qeq参数是合理的。具体的Qeq参数见下表。 分子1中ReaxFF和量子化学计算得到的原子电荷对比 分子2中ReaxFF和量子化学计算得到的原子电荷对比 分子3中ReaxFF和量子化学计算得到的原子电荷对比 ReaxFF模拟中采用的Qeq参数
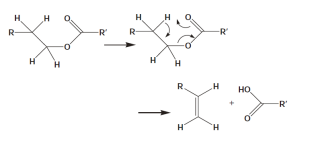
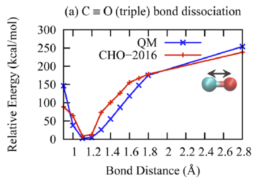
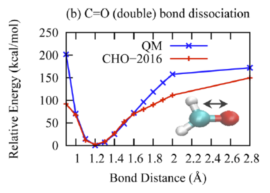
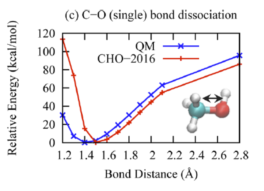
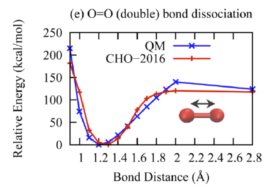
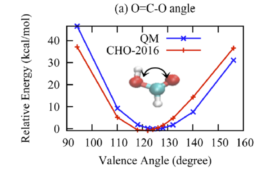
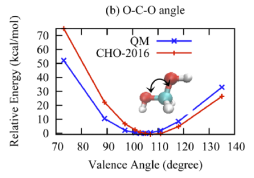
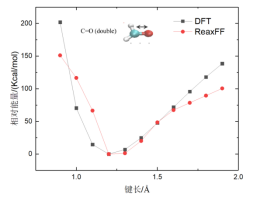
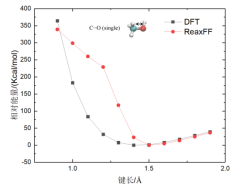
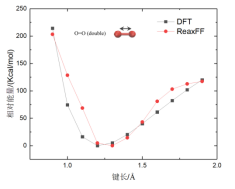
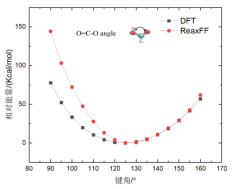
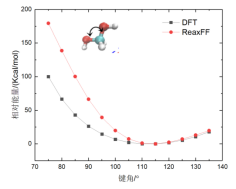
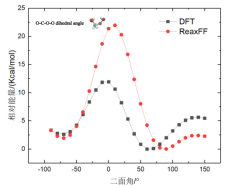
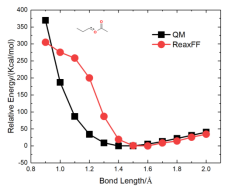
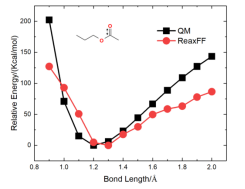
(2)键结能量验证方法 要验证的力场是针对CHO体系开发的。这里计划模拟合成酯,那么就要计算合成酯关键键的键能。前人研究表明合成酯在热解时会从CO单键处发生断键,所以需要重点验证这个键能。更一般的来说,当你找到一个力场,要针对自己的体系进行验证,你就需要将自己的体系中的分子进行简化,使其在包含关键化学基团的同时越简单越好。比如为了验证CO单键的能量这里选取了这个分子进行验证。首先验证关键的CO单键。验证的主要过程就是对比由DFT计算得到的能量和ReaxFF计算得到的能量,分子越简单能量对比越有效。为说明这里验证的方法的可行性(好像可行,后面会说明),首先复现一下论文中的结果。以下是论文Extension of the ReaxFF Combustion Force Field toward Syngas Combustion and Initial Oxidation Kinetics中的力场部分验证结果。
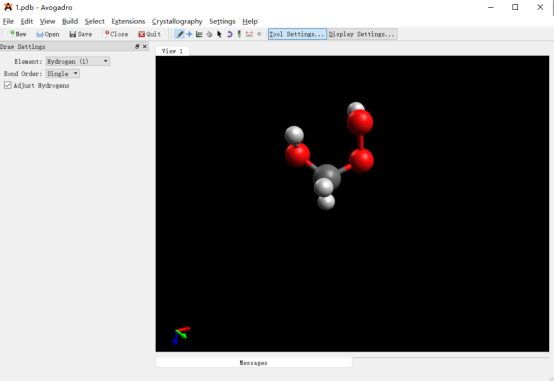
为了验证力场,首先要获得DFT的计算结果,也就是采用DFT方法对势能面性扫描。扫描采用高斯16软件进行刚性扫描。刚性扫描速度快,并且对于小分子来说柔性扫描和刚性扫描结果相差不大。下面通过上图中第六个图的结果说明高斯中如何进行刚性扫描。首先根据图中的分子在Avogadro软件中绘制出分子结构并保存为pdb文件。在绘制分子的时候尽量和图中的分子构象保持一致,如下图。
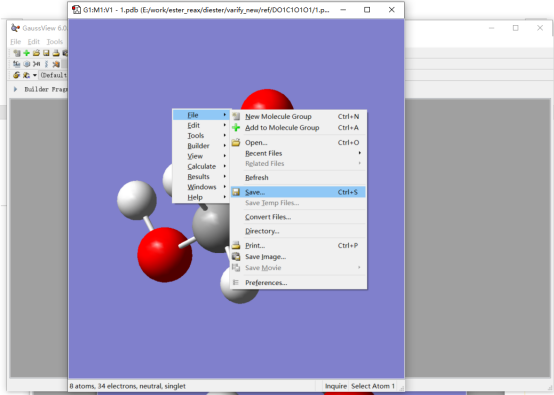
利用GaussView将保存的pdb文件打开,并保存为gif格式。保存的流程是在打开的窗口中鼠标右击选择File->Save…,打开保存窗口。
在打开的保存窗口中File name中设置文件名称,Files of type中选择Know files,取消Write Cartesians的勾选,然后点击保存。
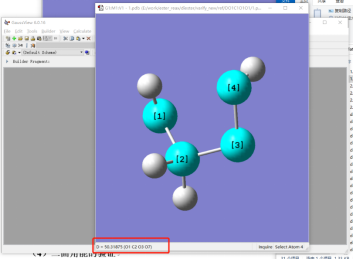
在刚才打开的pdb文件窗口中鼠标在原子上左击选择要扫描的二面角的四个原子。查看窗口左下角处构成该二面角的四个原子的编号。
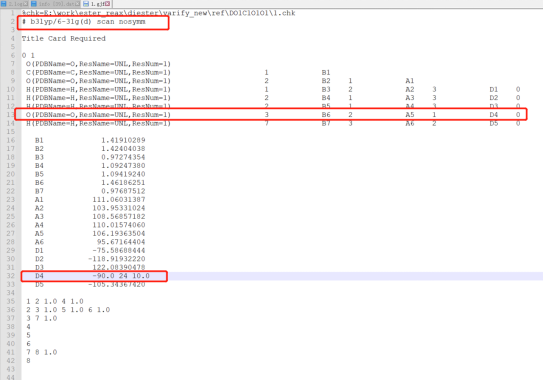
用文本编辑器将刚才保存的gif文件打开。将第二行改为b3lyp/6-31g(d) scan nosymm,其中b3lyp/6-31g(d)表示DFT计算要选择的基组,scan表示执行扫描任务,做刚性扫描的过程中,如果涉及到点群的改变,不加nosymm关键词的话有时会出问题,而且看到的扫描轨迹可能不连续。要扫描的二面角由编号为7 3 2 1四个原子组成,从文件中可以看出这个二面角的编号是D4。因此将下面D4行中的二面角角度改为-90.0 24 10.0。-90.0表示扫描初始角度为-90°,24表示扫描24个角度,10.0表示按10.0°进行角度递增。这里需要说明涉及到浮点数的一定要写为浮点数形式,比如角度要写为-90.0,10.0,而不能是-90和10。修改完后保存文件。
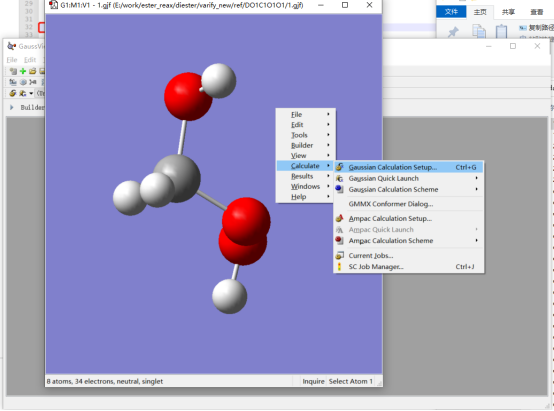
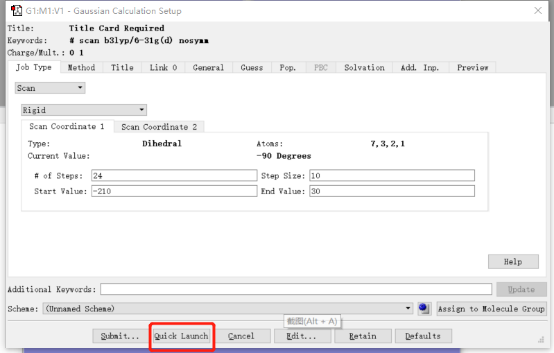
用GaussView打开刚刚修改并保存的gif文件,右击窗口空白处,按下图进行选择,打开计算设置窗口(不要关闭gif窗口)。
在打开的计算设置窗口中点击下方Quick Launch启动高斯开始计算。
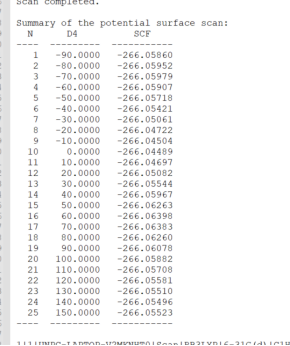
Quick Launch点击后会自动弹出高斯计算界面和另一个chk窗口。可以看到控制台在持续输出,由于是小分子,计算会比较快。计算完成后会弹出是否关闭高斯的对话框,点击是。稍等几秒钟,在chk窗口空白处鼠标右击,按下图进行选择打开保存临时文件窗口,在打开的窗口中设置保存的文件名称,点击保存。 会输出四个新的文件分别是chk,fch,gif,log文件。将log文件用文本编辑器打开,拉到文件末尾可以看到扫描得到的每个角度对应的能量,包括初始角度本例中会得到25行数据。将数据拷贝到绘图软件中。得到的能量的单位是Hartree,需要转换为Kcal/mol以和lammps的计算结果进行对比。1 Hartree=625.7 Kcal/mol。在对比DFT结果和ReaxFF结果时都会采用相对能量,即将所有计算得到的能量减去能量的最小值。
log文件中不仅会有扫描的最终结果,还会记录每一个角度对应的所有原子的坐标,通过提取这些坐标就可以得到每个角度对应的data文件,用以LAMMPS计算。提取log文件中的坐标并转换为一系列data文件的python脚本如下: work_path = 'E:\\work\\ester_reax\\diester\\varify_new\\ref\\DO1C1O1O1\\' #设置log文件的目录 input_file = open(work_path + '2.log','r')
natom = 8 #设置原子个数 line = input_file.readline() no = 0 while line: tmp1 = line.split() if len(tmp1) == 2: if tmp1[0] == "Z-Matrix" and tmp1[1] == "orientation:": no += 1 line = input_file.readline() line = input_file.readline() line = input_file.readline() line = input_file.readline() #data文件的头部,可按需修改,这里元素只有CHO三种 output_file = open(work_path + 'data.'+str(no),'w') output_file.write("LAMMPS data file Created\n") output_file.write("\n") output_file.write(str(natom) + " atoms\n") output_file.write("\n") output_file.write("3 atom types\n") output_file.write("\n") output_file.write("-20 20.0 xlo xhi\n") output_file.write("-20 20.0 ylo yhi\n") output_file.write("-20 20.0 zlo zhi\n") output_file.write("\n") output_file.write("Masses\n") output_file.write("\n") output_file.write("1 12.011\n") output_file.write("2 1.008\n") output_file.write("3 15.999\n") output_file.write("\n") output_file.write("Atoms\n") output_file.write("\n") for i in range(natom): line = input_file.readline().split() atom_id = line[0] mol_id = "1" #根据原子序号赋予LAMMPS中原子的type编号,可按需修改,这里只有CHO三种元素 if line[1] == "6": atom_type = "1" elif line[1] == "1": atom_type = "2" else: atom_type = "3" atom_q = "0" x = line[3] y = line[4] z = line[5] tmp = atom_id + " " + mol_id + " " + atom_type + " " + atom_q + " " + x + " " + y + " " + z + "\n" output_file.write(tmp) output_file.close() line = input_file.readline() 利用得到的一系列data文件就可以计算ReaxFF下的能量,计算的in文件如下: variable a loop 25 log log.$a variable b index 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 units real atom_style full neighbor 2.0 bin neigh_modify every 1 delay 0 check no read_data data.$b pair_style reaxff NULL pair_coeff * * ffield2016.reax.cho C H O timestep 0.25 velocity all create 300.0 4928459 rot yes dist gaussian thermo 100 thermo_style custom step temp pe ke etotal thermo_modify flush yes fix 3 all qeq/reaxff 1 0.0 10.0 1e-6 reaxff variable mye equal etotal run 1000 print ${mye} append info.dat screen no clear next b next a jump system.in 在输出的info.dat文件中就是所有data文件对应的能量,将数据拷贝到绘图软件中,并将所有数据减去这些能量中的最小值,得到相对能量。 由此便可对比DFT和ReaxFF的计算结果了。以上就是力场验证的所有过程。本例中对应文献中的六个图进行对比了DFT和ReaxFF的结果,如下图。从图中可以看出得到的对比图和文献中的差不多。但是还是有差别,特别是对于键来说,当键长小于平衡键长后这里计算的能量总是比文献中的较大,对于键角来说,当键角小于平衡键角后能量也比文献中的较大,对于二面角来说当二面角在零度左右也比文献中的大。但是对于ReaxFF和DFT对比的可接受程度来说,这里的验证是合理的。因为得到的误差和文献中的基本一致。至于为什么LAMMPS计算的ReaxFF的能量会与文献中的有符合很好的也有不符合的,我也百思不得其解。知道原因的可以加我微信baolu_yao,我们一起讨论。
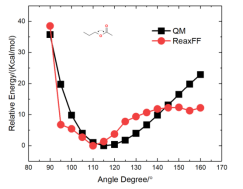
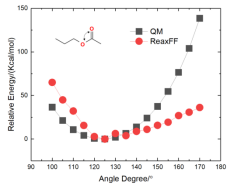
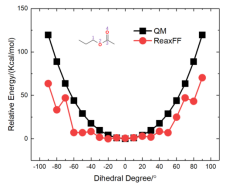
以下是针对合成酯分子的能量验证分别验证了酯基内的各类键,键角和二面角,不含酯基的部分不需要验证,力场本身已经在开发时验证过了。下面的结果表明包含酯基的ReaxFF计算的能量与DFT能量对比与原始力场相似,表明力场可以进行合成酯的模拟。 (3)键能的验证
(4)键角能的验证
(5)二面角能的验证
感谢鲍路瑶老师的分享,内容来自于鲍老师分享出来的资料 如有需要添加微信:lmp_zhushou 进入微信群,帮助他人,共建社区 获取完整版lammps讲义可以加微信lmp_zhushou或加入QQ群994359511 |