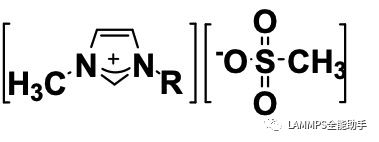|
LAMMPS讲解37-离子液体建模教程本例发布如何结合Ligpargen和moltemplate建立离子液体的in和data文件。这里以如下的离子液体为例
其中R=CH3。 首先在http://zarbi.chem.yale.edu/ligpargen/moleculeDraw.html这个网址的分子结构绘制面板中画出阳离子的结构,如下图
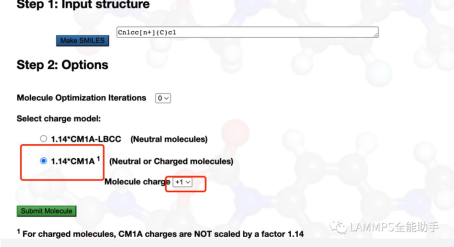
然后点击make smiles
然后按下图选择
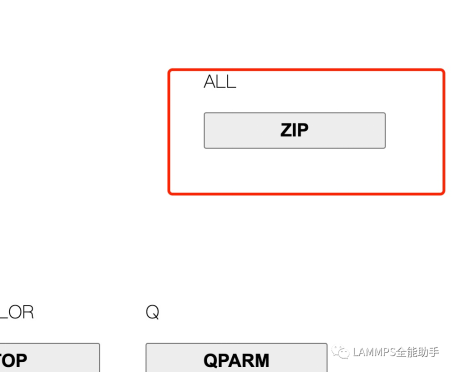
然后点击submit molecule,等待网站执行完成 然后点击ZIP下载文件
将下载文件解压缩后在解压出的文件找到后缀名为.lmp的文件,就是我们要的一个分子的data文件。 采用相同的方法建立阴离子的一个分子的data文件。注意建立阴离子需要将分子电荷选择为-1 将两个.lmp文件分别重命名为yanglizi.lmp和yinlizi.lmp,然后使用moltemplate的这个脚本ltemplify.py将data文件转换.lt文件 在装完moltemplate软件后这个脚本可以直接使用,在终端中进入yanglizi.lmp和yinlizi.lmp这两个文件所在的目录,然后分别执行以下两条命令 ltemplify.py -name yanglizi yanglizi.lmp > yanglizi.lt ltemplify.py -name yinlizi yinlizi.lmp > yinlizi.lt 由此就可得到阳离子和阴离子的.lt文件 然后就可以写system.lt文件对阴阳离子进行整列复制得到所需的data文件 import "yanglizi.lt" import "yinlizi.lt"
write_once("Data Boundary") { -50 550 xlo xhi -50 550 ylo yhi -50 550 zlo zhi } #移动间隔要根据阴阳离子的尺寸进行设定,如果不想麻烦就可以设定一个很大的值比如50 YAN = new yanglizi [10].move(0, 0, 50) [10].move(0, 50, 0) [10].move(50, 0, 0)
YIN = new yinlizi [10].move(0, 0, 50) [10].move(0, 50, 0) [10].move(50, 0, 0) #阴离子移动一下防止和阳离子重叠 YIN[*][*][*].move(50,50,50) 然后执行moltemplate.sh -atomstyle "full" system.lt,就可生成data文件了。in文件需要自己写,我们需要生成的system.data和system.in.settings文件。以下是弛豫示例的in文件。后续的分析可以在in文件中添加内容得到。 # ----------------- Init Section -----------------
boundary p p p neighbor 3.0 bin neigh_modify every 1 delay 0 check yes atom_style full units real bond_style harmonic angle_style harmonic dihedral_style opls improper_style cvff pair_style lj/cut/coul/long 8.5 8.5 pair_modify mix geometric special_bonds lj/coul 0.0 0.0 0.5 kspace_style pppm 0.0001
# ----------------- Atom Definition Section -----------------
read_data "system.data" include “system.in.settings”
# ----------------- Run Section -----------------
timestep 1.0
velocity all create 500 372748 rot yes dist gaussian loop local thermo 1000 thermo_style custom step temp thermo_modify flush yes
dump mydump all custom 10000 dump.lammpstrj id mol type mass x y z
fix fxnpt all npt temp 500 500 100.0 iso 100 100 1000
run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000 run 10000
unfix fxnpt fix fxnpt all npt temp 400 400 100.0 iso 1 1 1000 run 500000 write_data equ1.data write_restart restart1.equ
感谢鲍路瑶老师的分享,内容来自于鲍老师分享出来的资料 如有需要添加微信:lmp_zhushou 进入微信群,帮助他人,共建社区 获取完整版lammps讲义可以加微信lmp_zhushou或加入QQ群994359511 |