|
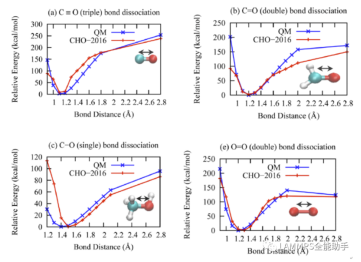
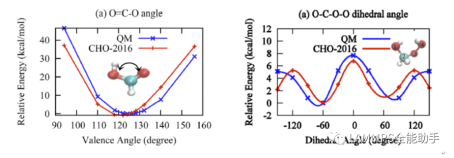
LAMMPS讲解34-ReaxFF力场之键结参数验证-对比DFT和ReaxFF当你找到一个力场,要针对自己的体系进行验证,你就需要将自己的体系中的分子进行简化,使其在包含关键化学基团的同时越简单越好。验证的主要过程就是对比由DFT计算得到的能量和ReaxFF计算得到的能量,分子越简单能量对比越有效。为说明这里验证的方法的可行性(好像可行,后面会说明),本例复现一下论文中的结果。以下是论文Extension of the ReaxFF Combustion Force Field toward Syngas Combustion and Initial Oxidation Kinetics中的力场部分验证结果。
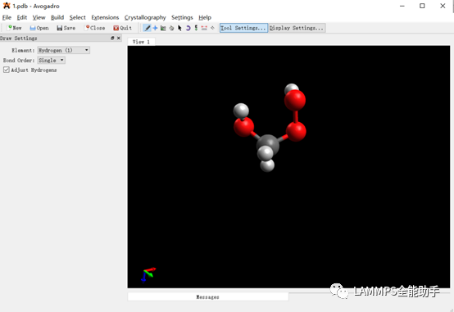
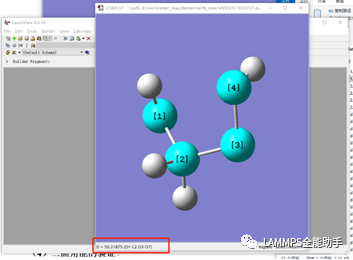
为了验证力场,首先要获得DFT的计算结果,也就是采用DFT方法对势能面性扫描。扫描采用高斯16软件进行刚性扫描。刚性扫描速度快,并且对于小分子来说柔性扫描和刚性扫描结果相差不大。下面通过上图中第六个图的结果说明高斯中如何进行刚性扫描。首先根据图中的分子在Avogadro软件中绘制出分子结构并保存为pdb文件。在绘制分子的时候尽量和图中的分子构象保持一致,如下图。
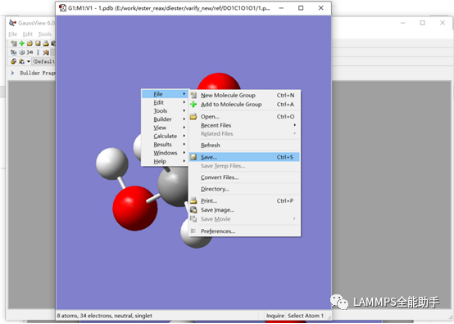
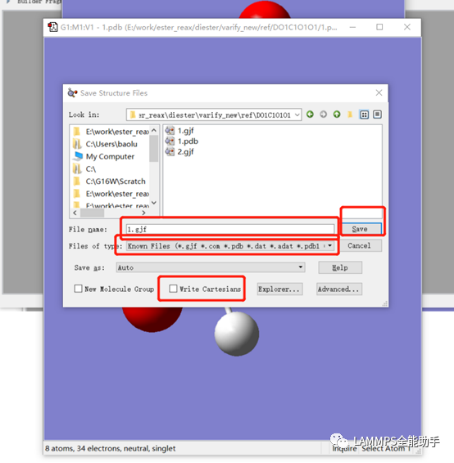
利用GaussView将保存的pdb文件打开,并保存为gif格式。保存的流程是在打开的窗口中鼠标右击选择File->Save…,打开保存窗口。
在打开的保存窗口中File name中设置文件名称,Files of type中选择Know files,取消Write Cartesians的勾选,然后点击保存。
在刚才打开的pdb文件窗口中鼠标在原子上左击选择要扫描的二面角的四个原子。查看窗口左下角处构成该二面角的四个原子的编号。
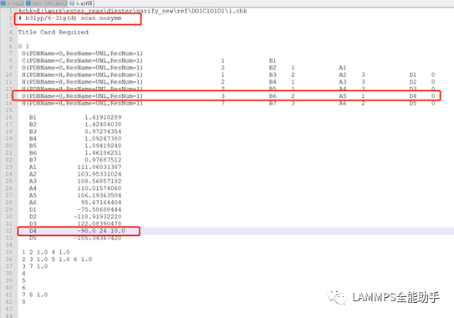
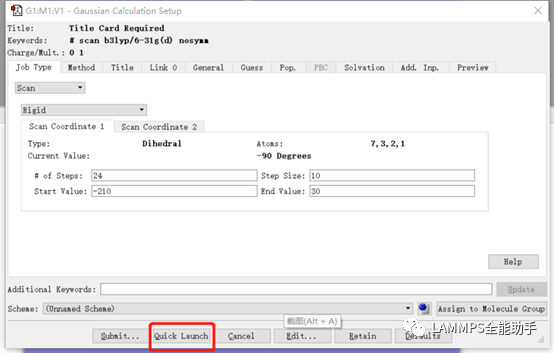
用文本编辑器将刚才保存的gif文件打开。将第二行改为b3lyp/6-31g(d) scan nosymm,其中b3lyp/6-31g(d)表示DFT计算要选择的基组,scan表示执行扫描任务,做刚性扫描的过程中,如果涉及到点群的改变,不加nosymm关键词的话有时会出问题,而且看到的扫描轨迹可能不连续。要扫描的二面角由编号为7 3 2 1四个原子组成,从文件中可以看出这个二面角的编号是D4。因此将下面D4行中的二面角角度改为-90.0 24 10.0。-90.0表示扫描初始角度为-90°,24表示扫描24个角度,10.0表示按10.0°进行角度递增。这里需要说明涉及到浮点数的一定要写为浮点数形式,比如角度要写为-90.0,10.0,而不能是-90和10。修改完后保存文件。
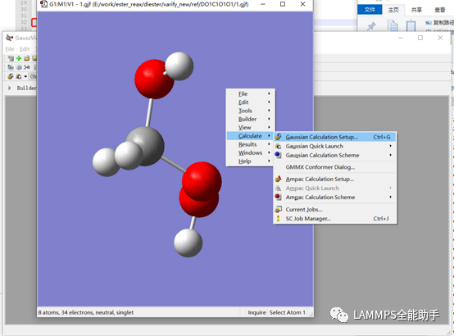
用GaussView打开刚刚修改并保存的gif文件,右击窗口空白处,按下图进行选择,打开计算设置窗口(不要关闭gif窗口)。
在打开的计算设置窗口中点击下方Quick Launch启动高斯开始计算。
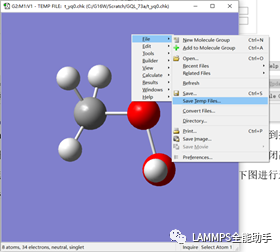
Quick Launch点击后会自动弹出高斯计算界面和另一个chk窗口。可以看到控制台在持续输出,由于是小分子,计算会比较快。计算完成后会弹出是否关闭高斯的对话框,点击是。稍等几秒钟,在chk窗口空白处鼠标右击,按下图进行选择打开保存临时文件窗口,在打开的窗口中设置保存的文件名称,点击保存。
会输出四个新的文件分别是chk,fch,gif,log文件。将log文件用文本编辑器打开,拉到文件末尾可以看到扫描得到的每个角度对应的能量,包括初始角度本例中会得到25行数据。将数据拷贝到绘图软件中。得到的能量的单位是Hartree,需要转换为Kcal/mol以和lammps的计算结果进行对比。1 Hartree=625.7 Kcal/mol。在对比DFT结果和ReaxFF结果时都会采用相对能量,即将所有计算得到的能量减去能量的最小值。
log文件中不仅会有扫描的最终结果,还会记录每一个角度对应的所有原子的坐标,通过提取这些坐标就可以得到每个角度对应的data文件,用以LAMMPS计算。提取log文件中的坐标并转换为一系列data文件的python脚本如下: #设置log文件的路径 work_path = 'E:\\work\\ester_reax\\diester\\varify_new\\ref\\DO1C1O1O1\\' input_file = open(work_path + '2.log','r') #设置原子个数 natom = 8 line = input_file.readline() no = 0 while line: tmp1 = line.split() if len(tmp1) == 2: if tmp1[0] == "Z-Matrix" and tmp1[1] == "orientation:": no += 1 line = input_file.readline() line = input_file.readline() line = input_file.readline() line = input_file.readline() output_file = open(work_path + 'data.'+str(no),'w') #LAMMPS的头部内容,可按需修改,这里只有CHO三种元素 output_file.write("LAMMPS data file Created\n") output_file.write("\n") output_file.write(str(natom) + " atoms\n") output_file.write("\n") output_file.write("3 atom types\n") output_file.write("\n") output_file.write("-20 20.0 xlo xhi\n") output_file.write("-20 20.0 ylo yhi\n") output_file.write("-20 20.0 zlo zhi\n") output_file.write("\n") output_file.write("Masses\n") output_file.write("\n") output_file.write("1 12.011\n") output_file.write("2 1.008\n") output_file.write("3 15.999\n") output_file.write("\n") output_file.write("Atoms\n") output_file.write("\n") for i in range(natom): line = input_file.readline().split() atom_id = line[0] mol_id = "1" #根据原子序号分配data文件中的原子type编号,这里只有CHO三种元素 if line[1] == "6": atom_type = "1" elif line[1] == "1": atom_type = "2" else: atom_type = "3" atom_q = "0" x = line[3] y = line[4] z = line[5] tmp = atom_id + " " + mol_id + " " + atom_type + " " + atom_q + " " + x + " " + y + " " + z + "\n" output_file.write(tmp) output_file.close() line = input_file.readline() 利用得到的一系列data文件就可以计算ReaxFF下的能量,计算的in文件如下: variable a loop 25 log log.$a variable b index 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 units real atom_style full neighbor 2.0 bin neigh_modify every 1 delay 0 check no read_data data.$b pair_style reaxff NULL pair_coeff * * ffield2016.reax.cho C H O timestep 0.25 velocity all create 300.0 4928459 rot yes dist gaussian thermo 100 thermo_style custom step temp pe ke etotal thermo_modify flush yes fix 3 all qeq/reaxff 1 0.0 10.0 1e-6 reaxff variable mye equal etotal run 1000 print ${mye} append info.dat screen no clear next b next a jump system.in 在输出的info.dat文件中就是所有data文件对应的能量,将数据拷贝到绘图软件中,并将所有数据减去这些能量中的最小值,得到相对能量。由此便可对比DFT和ReaxFF的计算结果了。以上就是力场验证的所有过程。本例中对应文献中的六个图进行对比了DFT和ReaxFF的结果,如下图。从图中可以看出得到的对比图和文献中的差不多。但是还是有差别,特别是对于键来说,当键长小于平衡键长后这里计算的能量总是比文献中的较大,对于键角来说,当键角小于平衡键角后能量也比文献中的较大,对于二面角来说当二面角在零度左右也比文献中的大。但是对于ReaxFF和DFT对比的可接受程度来说,这里的验证是合理的。因为得到的误差和文献中的基本一致。至于为什么LAMMPS计算的ReaxFF的能量会与文献中的有符合很好,也有不符合的,我也百思不得其解。知道原因的可以加我微信lmp_zhushou,我们一起讨论。
感谢鲍路瑶老师的分享,内容来自于鲍老师分享出来的资料 如有需要添加微信:lmp_zhushou 进入微信群,帮助他人,共建社区 获取完整版lammps讲义可以加微信lmp_zhushou或加入QQ群994359511
|