|
gmx_mmpbsa软件介绍简介
在Windows下使用时, 还需要一个bash环境, 你可以使用msys2, Git Bash, 或cygwin. 使用流程1. 获得输入文件
2. 设定 |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 文件 | 说明 |
|---|---|
gmx_mmpbsa.bsh | 总脚本 |
_pid.pdb | 转换为pdb的轨迹 |
_pid.qrv | 原子的电荷, 半径, VDW参数, 以及原子信息 |
| APBS相关文件 | |
_pid~XX.Yns.apbs | APBS输入文件 |
_pid~XX.Yns.out | APBS输出文件 |
_pid~XX.Yns_com.pqr | 复合物pqr文件 |
_pid~XX.Yns_lig.pqr | 配体pqr文件 |
_pid~XX.Yns_pro.pqr | 蛋白pqr文件 |
_pid~XX.Yns~COU+VDW.pdb | 复合物pdb文件, Occupancy列为COU能, beta列为VDW能 |
_pid~XX.Yns~PB+SA.pdb | 复合物pdb文件, Occupancy列为PB能, beta列为SA能 |
_pid~XX.Yns~res_MM+PBSA.pdb | 复合物pdb文件, Occupancy列为MM能, beta列为PBSA能 |
_pid~XX.Yns~res_MMPBSA.pdb | 复合物pdb文件, beta列为MMPBSA能 |
| 输出文件 | |
_pid~MMPBSA.dat | 总能量 |
_pid~res_MMPBSA.dat | 总能量分解到残基 |
_pid~resMM.dat | MM能量分解到残基 |
_pid~resMM_COU.dat | COU能量分解到残基 |
_pid~resMM_VDW.dat | VDW能量分解到残基 |
_pid~resPBSA.dat | PBSA能量分解到残基 |
_pid~resPBSA_PB.dat | PB能量分解到残基 |
_pid~resPBSA_SA.dat | SA能量分解到残基 |
测试: g_mmpbsa的1EBZ示例
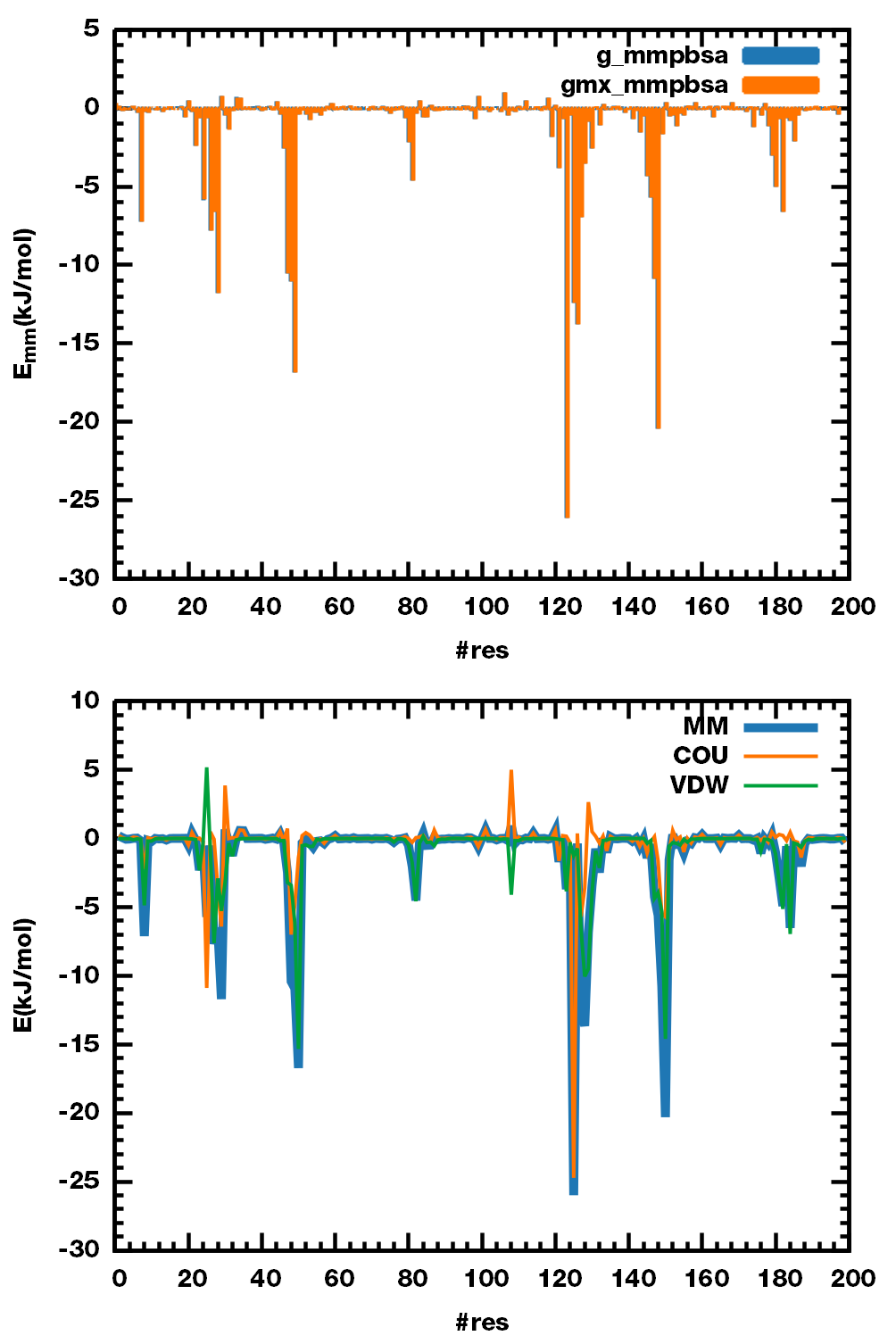
MM能量
计算方法: 直接对势累加, 考虑溶质(蛋白)的介电常数pdie, 不使用周期性, 不使用截断, 不对构型进行优化.
注意: GMXPBSA未考虑pdie, 且使用周期性, 使用截断, 对构型进行了优化, 但所得结果差距不大.
| Term | g_mmpbsa | gmx_mmpbsa | GMXPBSA |
|---|---|---|---|
| File | energy_MM.xvg | _pid~MMPBSA.dat | stru.rep |
| COU | -147.150 | -147.202 | -146.2175(-292.435/2) |
| VDW | -321.145 | -321.087 | -324.861 |
| MM(VDW+COU) | -468.295 | -468.289 | -471.0785 |
MM能量分解
计算方法: 直接累加
| Term | g_mmpbsa | gmx_mmpbsa | ||
|---|---|---|---|---|
| File | contrib_MM.dat | _pid~resMM.dat | _pid~resMM_COU.dat | _pid~resMM_VDW.dat |
| PRO-1 | 0.204 | 0.204 | 0.207 | -0.003 |
| GLN-2 | 0.061 | 0.061 | 0.066 | -0.005 |
| ILE-3 | -0.059 | -0.059 | -0.040 | -0.019 |
| THR-4 | -0.043 | -0.044 | -0.029 | -0.015 |
| LEU-5 | 0.002 | 0.002 | 0.091 | -0.090 |
| TRP-6 | 0.022 | 0.022 | 0.051 | -0.029 |
| GLN-7 | -0.169 | -0.169 | -0.113 | -0.056 |
| ARG-8 | -7.111 | -7.117 | -2.240 | -4.877 |
| PRO-9 | -0.139 | -0.139 | 0.039 | -0.179 |
| LEU-10 | -0.216 | -0.216 | -0.038 | -0.178 |
| VAL-11 | 0.001 | 0.001 | 0.033 | -0.032 |
| THR-12 | -0.035 | -0.035 | -0.025 | -0.010 |
| ILE-13 | -0.017 | -0.017 | 0.005 | -0.023 |
| LYS-14 | -0.118 | -0.118 | -0.113 | -0.005 |
| ILE-15 | -0.008 | -0.008 | 0.002 | -0.010 |
| GLY-16 | 0.002 | 0.002 | 0.004 | -0.001 |
| GLY-17 | -0.001 | -0.001 | -0.000 | -0.001 |
| GLN-18 | 0.003 | 0.003 | 0.007 | -0.004 |
| LEU-19 | 0.008 | 0.008 | 0.012 | -0.004 |
| LYS-20 | -0.500 | -0.499 | -0.482 | -0.017 |
| GLU-21 | 0.382 | 0.381 | 0.417 | -0.036 |
| ALA-22 | -0.136 | -0.135 | -0.052 | -0.083 |
| LEU-23 | -2.308 | -2.309 | 0.017 | -2.325 |
| LEU-24 | -0.536 | -0.536 | -0.245 | -0.291 |
| ASH-25 | -5.742 | -5.716 | -10.892 | 5.176 |
| THR-26 | -0.510 | -0.510 | 0.478 | -0.988 |
| GLY-27 | -7.689 | -7.685 | -0.059 | -7.626 |
| ALA-28 | -6.496 | -6.501 | -3.591 | -2.910 |
| ASP-29 | -11.702 | -11.700 | -6.418 | -5.282 |
| ASP-30 | 0.660 | 0.658 | 3.858 | -3.200 |
| THR-31 | -0.352 | -0.352 | -0.007 | -0.345 |
| VAL-32 | -1.243 | -1.244 | 0.053 | -1.297 |
| LEU-33 | -0.076 | -0.076 | 0.034 | -0.110 |
| GLU-34 | 0.577 | 0.576 | 0.618 | -0.042 |
| GLU-35 | 0.567 | 0.566 | 0.581 | -0.014 |
| MET-36 | -0.002 | -0.002 | 0.008 | -0.010 |
| SER-37 | -0.008 | -0.008 | -0.005 | -0.002 |
| LEU-38 | -0.007 | -0.007 | -0.002 | -0.006 |
| PRO-39 | 0.006 | 0.006 | 0.008 | -0.002 |
| GLY-40 | 0.011 | 0.011 | 0.012 | -0.001 |
| ARG-41 | -0.062 | -0.061 | -0.059 | -0.002 |
| TRP-42 | -0.020 | -0.020 | -0.012 | -0.007 |
| LYS-43 | 0.027 | 0.028 | 0.040 | -0.012 |
| PRO-44 | -0.045 | -0.045 | -0.034 | -0.011 |
| LYS-45 | 0.335 | 0.337 | 0.504 | -0.167 |
| MET-46 | -0.318 | -0.318 | -0.161 | -0.157 |
| ILE-47 | -2.494 | -2.493 | 0.717 | -3.210 |
| GLY-48 | -10.407 | -10.414 | -7.004 | -3.410 |
| GLY-49 | -10.918 | -10.921 | -4.801 | -6.120 |
| ILE-50 | -16.725 | -16.725 | -1.390 | -15.335 |
| GLY-51 | -0.328 | -0.327 | 0.202 | -0.530 |
| GLY-52 | -0.047 | -0.048 | 0.415 | -0.463 |
| PHE-53 | -0.314 | -0.313 | 0.249 | -0.563 |
| ILE-54 | -0.638 | -0.638 | -0.076 | -0.562 |
| LYS-55 | -0.215 | -0.213 | -0.158 | -0.055 |
| VAL-56 | -0.171 | -0.171 | -0.045 | -0.127 |
| ARG-57 | -0.372 | -0.371 | -0.347 | -0.024 |
| GLN-58 | -0.067 | -0.067 | -0.038 | -0.029 |
| TYR-59 | 0.016 | 0.016 | 0.029 | -0.013 |
| ASP-60 | 0.203 | 0.202 | 0.209 | -0.007 |
| GLN-61 | -0.028 | -0.028 | -0.024 | -0.004 |
| ILE-62 | -0.018 | -0.018 | -0.012 | -0.006 |
| LEU-63 | 0.009 | 0.009 | 0.012 | -0.003 |
| ILE-64 | -0.030 | -0.030 | -0.017 | -0.013 |
| GLU-65 | 0.055 | 0.055 | 0.060 | -0.005 |
| ILE-66 | -0.022 | -0.022 | -0.003 | -0.018 |
| CYS-67 | -0.015 | -0.015 | -0.007 | -0.008 |
| GLY-68 | -0.006 | -0.006 | -0.004 | -0.002 |
| HIS-69 | -0.000 | -0.000 | 0.004 | -0.004 |
| LYS-70 | -0.054 | -0.054 | -0.051 | -0.003 |
| ALA-71 | 0.008 | 0.008 | 0.012 | -0.003 |
| ILE-72 | -0.016 | -0.016 | -0.011 | -0.005 |
| GLY-73 | -0.024 | -0.024 | -0.019 | -0.005 |
| THR-74 | 0.010 | 0.010 | 0.042 | -0.033 |
| VAL-75 | -0.083 | -0.082 | -0.048 | -0.035 |
| LEU-76 | -0.276 | -0.276 | -0.011 | -0.265 |
| VAL-77 | -0.057 | -0.057 | -0.020 | -0.037 |
| GLY-78 | -0.028 | -0.028 | 0.003 | -0.031 |
| PRO-79 | 0.040 | 0.040 | 0.126 | -0.086 |
| THR-80 | -0.507 | -0.507 | -0.011 | -0.496 |
| PRO-81 | -2.052 | -2.051 | -0.282 | -1.769 |
| VAL-82 | -4.520 | -4.520 | 0.056 | -4.576 |
| ASN-83 | -0.244 | -0.244 | 0.097 | -0.341 |
| ILE-84 | 0.383 | 0.376 | 0.220 | 0.156 |
| ILE-85 | -0.503 | -0.503 | -0.207 | -0.296 |
| GLY-86 | -0.489 | -0.489 | -0.223 | -0.266 |
| ARG-87 | 0.074 | 0.070 | 0.606 | -0.536 |
| ASN-88 | -0.087 | -0.086 | 0.025 | -0.111 |
| LEU-89 | -0.051 | -0.051 | -0.010 | -0.041 |
| LEU-90 | -0.048 | -0.048 | 0.008 | -0.056 |
| THR-91 | -0.006 | -0.006 | 0.010 | -0.016 |
| GLN-92 | -0.035 | -0.035 | -0.026 | -0.010 |
| ILE-93 | -0.006 | -0.006 | 0.003 | -0.009 |
| GLY-94 | -0.010 | -0.010 | -0.007 | -0.003 |
| CYS-95 | -0.041 | -0.041 | -0.029 | -0.012 |
| THR-96 | 0.058 | 0.058 | 0.069 | -0.011 |
| LEU-97 | 0.038 | 0.038 | 0.087 | -0.049 |
| ASN-98 | -0.093 | -0.093 | -0.084 | -0.010 |
| PHE-99 | -0.609 | -0.609 | -0.596 | -0.013 |
| PRO-101 | 0.637 | 0.637 | 0.641 | -0.004 |
| GLN-102 | -0.006 | -0.006 | -0.002 | -0.005 |
| ILE-103 | -0.007 | -0.007 | 0.012 | -0.018 |
| THR-104 | -0.107 | -0.107 | -0.095 | -0.012 |
| LEU-105 | -0.009 | -0.009 | 0.055 | -0.064 |
| TRP-106 | 0.005 | 0.006 | 0.038 | -0.032 |
| GLN-107 | -0.135 | -0.135 | -0.085 | -0.050 |
| ARG-108 | 0.903 | 0.895 | 4.998 | -4.102 |
| PRO-109 | -0.350 | -0.350 | -0.193 | -0.156 |
| LEU-110 | -0.072 | -0.072 | 0.090 | -0.161 |
| VAL-111 | -0.107 | -0.107 | -0.077 | -0.031 |
| THR-112 | 0.026 | 0.026 | 0.035 | -0.010 |
| ILE-113 | -0.016 | -0.016 | 0.011 | -0.027 |
| LYS-114 | 0.392 | 0.391 | 0.396 | -0.005 |
| ILE-115 | -0.019 | -0.019 | -0.007 | -0.012 |
| GLY-116 | -0.005 | -0.005 | -0.003 | -0.002 |
| GLY-117 | 0.003 | 0.003 | 0.004 | -0.001 |
| GLN-118 | -0.055 | -0.055 | -0.050 | -0.005 |
| LEU-119 | 0.014 | 0.014 | 0.019 | -0.005 |
| LYS-120 | 0.558 | 0.558 | 0.579 | -0.022 |
| GLU-121 | -1.704 | -1.704 | -1.638 | -0.065 |
| ALA-122 | 0.102 | 0.102 | 0.200 | -0.098 |
| LEU-123 | -3.696 | -3.696 | 0.157 | -3.853 |
| LEU-124 | -0.594 | -0.594 | -0.302 | -0.292 |
| ASP-125 | -25.997 | -25.986 | -24.723 | -1.263 |
| THR-126 | -0.361 | -0.361 | 0.367 | -0.728 |
| GLY-127 | -12.305 | -12.314 | -5.690 | -6.624 |
| ALA-128 | -13.661 | -13.660 | -3.604 | -10.056 |
| ASP-129 | -6.844 | -6.849 | 2.647 | -9.495 |
| ASP-130 | -3.428 | -3.433 | 0.495 | -3.928 |
| THR-131 | -0.750 | -0.750 | 0.150 | -0.901 |
| VAL-132 | -2.478 | -2.477 | -0.331 | -2.147 |
| LEU-133 | 0.007 | 0.007 | 0.162 | -0.156 |
| GLU-134 | -1.015 | -1.015 | -0.903 | -0.111 |
| GLU-135 | 0.130 | 0.130 | 0.153 | -0.022 |
| MET-136 | -0.017 | -0.017 | -0.002 | -0.015 |
| SER-137 | -0.029 | -0.029 | -0.025 | -0.004 |
| LEU-138 | 0.008 | 0.008 | 0.016 | -0.008 |
| PRO-139 | 0.009 | 0.010 | 0.012 | -0.002 |
| GLY-140 | -0.003 | -0.003 | -0.002 | -0.001 |
| ARG-141 | -0.200 | -0.200 | -0.198 | -0.003 |
| TRP-142 | -0.008 | -0.008 | 0.004 | -0.012 |
| LYS-143 | -0.574 | -0.574 | -0.559 | -0.016 |
| PRO-144 | -0.056 | -0.056 | -0.040 | -0.015 |
| LYS-145 | -1.415 | -1.415 | -1.068 | -0.347 |
| MET-146 | -0.406 | -0.407 | -0.249 | -0.157 |
| ILE-147 | -4.248 | -4.249 | 0.125 | -4.374 |
| GLY-148 | -5.601 | -5.602 | -1.655 | -3.947 |
| GLY-149 | -10.792 | -10.785 | -5.451 | -5.334 |
| ILE-150 | -20.312 | -20.303 | -5.702 | -14.601 |
| GLY-151 | -1.550 | -1.551 | -0.968 | -0.583 |
| GLY-152 | 0.274 | 0.274 | 0.580 | -0.306 |
| PHE-153 | -0.435 | -0.436 | -0.082 | -0.354 |
| ILE-154 | -0.386 | -0.386 | 0.102 | -0.488 |
| LYS-155 | -1.053 | -1.053 | -0.987 | -0.066 |
| VAL-156 | -0.268 | -0.268 | 0.006 | -0.274 |
| ARG-157 | -0.385 | -0.385 | -0.337 | -0.048 |
| GLN-158 | 0.051 | 0.051 | 0.102 | -0.051 |
| TYR-159 | 0.008 | 0.008 | 0.030 | -0.023 |
| ASP-160 | 0.282 | 0.282 | 0.292 | -0.011 |
| GLN-161 | -0.009 | -0.009 | -0.005 | -0.004 |
| ILE-162 | -0.013 | -0.013 | -0.004 | -0.009 |
| LEU-163 | -0.005 | -0.005 | -0.000 | -0.004 |
| ILE-164 | 0.019 | 0.019 | 0.034 | -0.015 |
| GLU-165 | -0.459 | -0.459 | -0.454 | -0.005 |
| ILE-166 | 0.030 | 0.030 | 0.047 | -0.018 |
| CYS-167 | -0.000 | -0.000 | 0.005 | -0.006 |
| GLY-168 | 0.023 | 0.023 | 0.025 | -0.001 |
| HIS-169 | 0.008 | 0.008 | 0.012 | -0.004 |
| LYS-170 | 0.240 | 0.239 | 0.243 | -0.003 |
| ALA-171 | -0.017 | -0.017 | -0.012 | -0.005 |
| ILE-172 | -0.005 | -0.005 | 0.001 | -0.006 |
| GLY-173 | -0.014 | -0.014 | -0.006 | -0.008 |
| THR-174 | -0.119 | -0.119 | -0.060 | -0.058 |
| VAL-175 | 0.005 | 0.005 | 0.063 | -0.058 |
| LEU-176 | -1.081 | -1.080 | -0.139 | -0.942 |
| VAL-177 | -0.005 | -0.005 | 0.070 | -0.075 |
| GLY-178 | -0.343 | -0.343 | -0.268 | -0.075 |
| PRO-179 | 0.234 | 0.234 | 0.466 | -0.232 |
| THR-180 | -1.020 | -1.020 | 0.084 | -1.104 |
| PRO-181 | -2.907 | -2.907 | 0.285 | -3.191 |
| VAL-182 | -4.923 | -4.922 | 0.213 | -5.135 |
| ASN-183 | -0.565 | -0.565 | -0.152 | -0.413 |
| ILE-184 | -6.518 | -6.519 | 0.443 | -6.961 |
| ILE-185 | -0.517 | -0.517 | -0.183 | -0.334 |
| GLY-186 | -0.707 | -0.708 | -0.281 | -0.427 |
| ARG-187 | -2.021 | -2.020 | -1.384 | -0.636 |
| ASN-188 | -0.358 | -0.358 | -0.161 | -0.198 |
| LEU-189 | -0.012 | -0.012 | 0.051 | -0.063 |
| LEU-190 | -0.077 | -0.077 | 0.036 | -0.113 |
| THR-191 | -0.044 | -0.044 | -0.020 | -0.024 |
| GLN-192 | -0.011 | -0.011 | 0.001 | -0.012 |
| ILE-193 | 0.008 | 0.008 | 0.024 | -0.015 |
| GLY-194 | -0.010 | -0.010 | -0.006 | -0.004 |
| CYS-195 | -0.047 | -0.047 | -0.026 | -0.021 |
| THR-196 | -0.040 | -0.040 | -0.028 | -0.012 |
| LEU-197 | 0.010 | 0.010 | 0.058 | -0.048 |
| ASN-198 | 0.027 | 0.027 | 0.034 | -0.007 |
| PHE-199 | -0.295 | -0.295 | -0.287 | -0.007 |
| BEC-200 | -234.069 | -234.145 | -73.601 | -160.544 |
PB(极性)能量, Polar solvation enrgy
计算方法: 水相能量减去真空中的能量, PB=Esol-Evac
| Term | com | pro | lig |
|---|---|---|---|
| Esol | 164517.154959 | 161287.089351 | 1841.194271 |
| Evac | 170135.930761 | 167060.999097 | 1997.546166 |
| PB | -5618.775802 | -5773.909746 | -156.351895 |
| Term | g_mmpbsa | gmx_mmpbsa (相同电荷/网格) | gmx_mmpbsa | GMXPBSA |
|---|---|---|---|---|
| File | polar.xvg | _pid~MMPBSA.dat | stru0.out | |
| com | -5618.776 | -5658.630 | -5786.034 | -8830.573 |
| pro | -5773.910 | -5816.675 | -5955.618 | -9038.944 |
| lig | -156.352 | -152.874 | -153.638 | -166.433 |
| dPB(com-pro-lig) | 311.486 | 310.919 | 323.222 | 374.805 |
与g_mmpbsa比较, PB能量有差距, 原因在于
所用网格不同,
g_mmpbsa对每个分子构型使用基于其自身的网格, 而gmx_mmpbsa使用基于所有构型的网格, 所有APBS计算使用相同的网格.有些原子的半径不同,
g_mmpbsa根据蛋白原子名称推断半径的方式有些怪异, 与论文所给数据不一致. 对配体, 由于原子名称可以任意取, 所以没有办法直接根据名称推断半径. 下面是g_mmpbsa所给蛋白中原子的半径, 可以看到有很多不一致之处.CA 1.7 CB 1.7 CD 1.7 CD1 1.7 CD1 1.77 CD2 1.7 CD2 1.77 CE 1.7 CE1 1.77 CE2 1.77 CE3 1.77 CG 1.7 CG 1.77 CG1 1.7 CG2 1.7 CH2 1.77 CZ 1.7 CZ 1.77 CZ2 1.77 CZ3 1.77 H 1.2 H1 1.2 H2 1.2 HA 1.2 HA1 1.2 HA2 1.2 HB 1.2 HB1 1.2 HB2 1.2 HB3 1.2 HD1 1. HD1 1.2 HD11 1.2 HD12 1.2 HD13 1.2 HD2 1. HD2 1.2 HD21 1.2 HD22 1.2 HD23 1.2 HD3 1.2 HE 1.2 HE1 1. HE1 1.2 HE2 1. HE2 1.2 HE21 1.2 HE22 1.2 HE3 1. HE3 1.2 HG 1.2 HG1 1.2 HG11 1.2 HG12 1.2 HG13 1.2 HG2 1.2 HG21 1.2 HG22 1.2 HG23 1.2 HH 1.2 HH11 1.2 HH12 1.2 HH2 1. HH21 1.2 HH22 1.2 HZ 1. HZ1 1.2 HZ2 1. HZ2 1.2 HZ3 1. HZ3 1.2
换用与
g_mmpbsa相同的网格与半径后, 所得PB作用能差距很小.GMXPBSA所用网格更大, 且使用了来自LJ参数的半径, 并对构型进行了优化, 所得结果也更大.
PB能量分解
计算方法: 残基处于复合物中时的能量减去处于其所属分子(蛋白/配体)中的能量, G(res@com)-G(res@pro)
| Term | PRO-1@com | PRO-1@pro | lig@com | lig@lig |
|---|---|---|---|---|
| sol | 453.312984 | 453.322869 | 3197.166762 | 1841.194271 |
| vac | 490.849104 | 490.694707 | 3213.732072 | 1997.546166 |
| PB | -37.53612 | -37.371838 | -16.56531 | -156.351895 |
| dPB(@com-@pro) | -0.164282 | 139.786585 | ||
| Term | g_mmpbsa | gmx_mmpbsa |
|---|---|---|
| File | contrib_pol.dat | _pid~resPBSA_PB.dat |
| PRO-1 | -0.164 | -0.151 |
| GLN-2 | -0.120 | -0.120 |
| ILE-3 | 0.090 | 0.094 |
| THR-4 | 0.111 | 0.109 |
| LEU-5 | -0.073 | -0.070 |
| TRP-6 | 0.024 | 0.024 |
| GLN-7 | 0.209 | 0.212 |
| ARG-8 | 13.074 | 13.140 |
| PRO-9 | -0.051 | -0.059 |
| LEU-10 | 0.050 | 0.060 |
| VAL-11 | -0.031 | -0.033 |
| THR-12 | -0.001 | -0.001 |
| ILE-13 | -0.021 | -0.019 |
| LYS-14 | 0.047 | 0.058 |
| ILE-15 | 0.002 | 0.003 |
| GLY-16 | 0.008 | 0.009 |
| GLY-17 | 0.006 | 0.008 |
| GLN-18 | 0.009 | 0.009 |
| LEU-19 | -0.008 | -0.006 |
| LYS-20 | 0.283 | 0.311 |
| GLU-21 | -0.184 | -0.210 |
| ALA-22 | -0.016 | -0.006 |
| LEU-23 | 0.115 | 0.128 |
| LEU-24 | 0.491 | 0.500 |
| ASH-25 | 6.903 | 6.918 |
| THR-26 | -0.757 | -0.812 |
| GLY-27 | 6.913 | 8.089 |
| ALA-28 | 2.982 | 3.538 |
| ASP-29 | 9.555 | 9.422 |
| ASP-30 | 4.460 | 5.257 |
| THR-31 | 0.005 | 0.008 |
| VAL-32 | 0.197 | 0.176 |
| LEU-33 | -0.062 | -0.060 |
| GLU-34 | -0.377 | -0.415 |
| GLU-35 | -0.339 | -0.355 |
| MET-36 | -0.018 | -0.019 |
| SER-37 | 0.006 | 0.007 |
| LEU-38 | 0.005 | 0.006 |
| PRO-39 | -0.021 | -0.023 |
| GLY-40 | -0.013 | -0.014 |
| ARG-41 | 0.012 | 0.013 |
| TRP-42 | 0.018 | 0.019 |
| LYS-43 | 0.165 | 0.180 |
| PRO-44 | 0.032 | 0.034 |
| LYS-45 | 0.841 | 0.938 |
| MET-46 | 0.236 | 0.236 |
| ILE-47 | 0.328 | 0.367 |
| GLY-48 | 13.029 | 14.797 |
| GLY-49 | 7.420 | 7.726 |
| ILE-50 | 1.984 | 1.969 |
| GLY-51 | 0.212 | 0.215 |
| GLY-52 | -0.763 | -0.778 |
| PHE-53 | -0.124 | -0.133 |
| ILE-54 | -0.141 | -0.152 |
| LYS-55 | 0.335 | 0.353 |
| VAL-56 | 0.049 | 0.052 |
| ARG-57 | 0.383 | 0.406 |
| GLN-58 | -0.055 | -0.057 |
| TYR-59 | -0.032 | -0.036 |
| ASP-60 | -0.297 | -0.328 |
| GLN-61 | 0.013 | 0.017 |
| ILE-62 | 0.016 | 0.015 |
| LEU-63 | -0.020 | -0.021 |
| ILE-64 | -0.006 | -0.003 |
| GLU-65 | -0.011 | -0.030 |
| ILE-66 | -0.034 | -0.030 |
| CYS-67 | 0.004 | 0.006 |
| GLY-68 | -0.007 | -0.005 |
| HIS-69 | 0.028 | 0.026 |
| LYS-70 | 0.027 | 0.036 |
| ALA-71 | -0.001 | -0.000 |
| ILE-72 | 0.006 | 0.006 |
| GLY-73 | 0.011 | 0.014 |
| THR-74 | 0.035 | 0.033 |
| VAL-75 | -0.028 | -0.027 |
| LEU-76 | 0.094 | 0.088 |
| VAL-77 | -0.022 | -0.024 |
| GLY-78 | 0.053 | 0.049 |
| PRO-79 | -0.164 | -0.162 |
| THR-80 | 0.006 | 0.010 |
| PRO-81 | 0.412 | 0.451 |
| VAL-82 | 0.187 | 0.214 |
| ASN-83 | -0.132 | -0.157 |
| ILE-84 | -0.198 | -0.180 |
| ILE-85 | 0.413 | 0.410 |
| GLY-86 | -0.097 | -0.089 |
| ARG-87 | -2.641 | -2.455 |
| ASN-88 | 0.299 | 0.305 |
| LEU-89 | -0.127 | -0.124 |
| LEU-90 | -0.136 | -0.131 |
| THR-91 | -0.018 | -0.016 |
| GLN-92 | -0.049 | -0.044 |
| ILE-93 | -0.033 | -0.030 |
| GLY-94 | -0.020 | -0.016 |
| CYS-95 | 0.046 | 0.040 |
| THR-96 | -0.091 | -0.092 |
| LEU-97 | -0.259 | -0.259 |
| ASN-98 | 0.301 | 0.303 |
| PHE-99 | 0.566 | 0.550 |
| PRO-101 | -0.397 | -0.386 |
| GLN-102 | -0.075 | -0.076 |
| ILE-103 | 0.061 | 0.064 |
| THR-104 | 0.098 | 0.096 |
| LEU-105 | -0.026 | -0.038 |
| TRP-106 | 0.003 | -0.006 |
| GLN-107 | 0.136 | 0.132 |
| ARG-108 | 2.167 | 2.654 |
| PRO-109 | 0.336 | 0.333 |
| LEU-110 | -0.128 | -0.126 |
| VAL-111 | 0.117 | 0.116 |
| THR-112 | -0.050 | -0.051 |
| ILE-113 | -0.121 | -0.119 |
| LYS-114 | -0.172 | -0.167 |
| ILE-115 | -0.055 | -0.054 |
| GLY-116 | -0.006 | -0.005 |
| GLY-117 | -0.021 | -0.021 |
| GLN-118 | 0.056 | 0.055 |
| LEU-119 | -0.041 | -0.041 |
| LYS-120 | -0.300 | -0.281 |
| GLU-121 | 0.584 | 0.556 |
| ALA-122 | -0.406 | -0.411 |
| LEU-123 | 0.498 | 0.551 |
| LEU-124 | 0.220 | 0.210 |
| ASP-125 | 51.872 | 55.384 |
| THR-126 | -1.119 | -1.122 |
| GLY-127 | 11.324 | 12.291 |
| ALA-128 | 4.202 | 4.744 |
| ASP-129 | 2.239 | 1.697 |
| ASP-130 | 13.444 | 15.504 |
| THR-131 | -0.439 | -0.429 |
| VAL-132 | 0.663 | 0.677 |
| LEU-133 | -0.119 | -0.118 |
| GLU-134 | 0.372 | 0.341 |
| GLU-135 | -0.172 | -0.186 |
| MET-136 | 0.091 | 0.091 |
| SER-137 | 0.016 | 0.013 |
| LEU-138 | -0.028 | -0.029 |
| PRO-139 | 0.008 | 0.007 |
| GLY-140 | -0.001 | -0.001 |
| ARG-141 | 0.021 | 0.022 |
| TRP-142 | -0.017 | -0.018 |
| LYS-143 | 0.156 | 0.169 |
| PRO-144 | 0.064 | 0.065 |
| LYS-145 | -0.483 | -0.422 |
| MET-146 | 0.366 | 0.357 |
| ILE-147 | 0.804 | 0.763 |
| GLY-148 | 9.259 | 9.576 |
| GLY-149 | 6.130 | 7.090 |
| ILE-150 | 4.967 | 4.954 |
| GLY-151 | 1.110 | 1.124 |
| GLY-152 | -0.613 | -0.606 |
| PHE-153 | 0.121 | 0.111 |
| ILE-154 | -0.125 | -0.136 |
| LYS-155 | 0.578 | 0.587 |
| VAL-156 | -0.097 | -0.093 |
| ARG-157 | 0.315 | 0.333 |
| GLN-158 | -0.218 | -0.233 |
| TYR-159 | 0.076 | 0.073 |
| ASP-160 | 0.124 | 0.111 |
| GLN-161 | -0.052 | -0.051 |
| ILE-162 | 0.047 | 0.047 |
| LEU-163 | 0.023 | 0.022 |
| ILE-164 | -0.063 | -0.061 |
| GLU-165 | 0.324 | 0.316 |
| ILE-166 | -0.075 | -0.071 |
| CYS-167 | 0.030 | 0.030 |
| GLY-168 | -0.041 | -0.041 |
| HIS-169 | 0.004 | 0.004 |
| LYS-170 | -0.148 | -0.144 |
| ALA-171 | -0.003 | -0.005 |
| ILE-172 | -0.033 | -0.032 |
| GLY-173 | -0.034 | -0.033 |
| THR-174 | 0.280 | 0.283 |
| VAL-175 | -0.314 | -0.317 |
| LEU-176 | 0.161 | 0.155 |
| VAL-177 | -0.067 | -0.074 |
| GLY-178 | 0.274 | 0.275 |
| PRO-179 | -0.595 | -0.604 |
| THR-180 | 0.535 | 0.635 |
| PRO-181 | -0.029 | 0.015 |
| VAL-182 | 0.655 | 0.636 |
| ASN-183 | 0.158 | 0.144 |
| ILE-184 | -0.982 | -0.997 |
| ILE-185 | 0.336 | 0.354 |
| GLY-186 | -0.727 | -0.728 |
| ARG-187 | -0.185 | -0.107 |
| ASN-188 | 0.616 | 0.630 |
| LEU-189 | -0.417 | -0.426 |
| LEU-190 | -0.402 | -0.407 |
| THR-191 | -0.018 | -0.017 |
| GLN-192 | -0.171 | -0.175 |
| ILE-193 | -0.154 | -0.151 |
| GLY-194 | -0.065 | -0.064 |
| CYS-195 | 0.133 | 0.128 |
| THR-196 | 0.005 | 0.005 |
| LEU-197 | -0.160 | -0.161 |
| ASN-198 | -0.088 | -0.087 |
| PHE-199 | 0.229 | 0.211 |
| BEC-200 | 139.787 | 138.191 |
SA(非极性)能量, APolar solvation energy
计算方法: 每个原子的溶剂可及表面积乘以表面张力, 加上常量
| Term | g_mmpbsa | gmx_mmpbsa | GMXPBSA |
|---|---|---|---|
| File | sasa.dat | _pid~resPBSA_SA.dat | Hstru0.out |
| com | 232.144 | 229.960 | 223.818 |
| pro | 243.428 | 240.034 | 233.557 |
| lig | 21.155 | 24.586 | 21.243 |
| dSA(com-pro-lig) | -32.439 | -34.660 | -30.982 |
SA能量分解
计算方法: 直接累加
| Term | g_mmpbsa | gmx_mmpbsa |
|---|---|---|
| File | sasa_contrib.dat | _pid~resPBSA_SA.dat |
| PRO-1 | 0.000 | -0.001 |
| GLN-2 | 0.000 | -0.001 |
| ILE-3 | 0.000 | -0.001 |
| THR-4 | 0.000 | -0.000 |
| LEU-5 | 0.000 | -0.001 |
| TRP-6 | 0.000 | -0.001 |
| GLN-7 | 0.000 | -0.001 |
| ARG-8 | -0.847 | -0.703 |
| PRO-9 | 0.000 | -0.000 |
| LEU-10 | 0.000 | -0.001 |
| VAL-11 | 0.000 | -0.001 |
| THR-12 | 0.000 | -0.000 |
| ILE-13 | 0.000 | -0.001 |
| LYS-14 | 0.000 | -0.001 |
| ILE-15 | 0.000 | -0.001 |
| GLY-16 | 0.000 | -0.000 |
| GLY-17 | 0.000 | -0.000 |
| GLN-18 | 0.000 | -0.001 |
| LEU-19 | 0.000 | -0.001 |
| LYS-20 | 0.000 | -0.001 |
| GLU-21 | 0.000 | -0.001 |
| ALA-22 | 0.000 | -0.000 |
| LEU-23 | -0.206 | -0.138 |
| LEU-24 | 0.000 | -0.001 |
| ASH-25 | -0.332 | -0.280 |
| THR-26 | 0.000 | -0.000 |
| GLY-27 | -0.440 | -0.474 |
| ALA-28 | -0.482 | -0.403 |
| ASP-29 | -0.332 | -0.471 |
| ASP-30 | -0.272 | -0.251 |
| THR-31 | 0.000 | -0.000 |
| VAL-32 | -0.060 | -0.098 |
| LEU-33 | 0.000 | -0.001 |
| GLU-34 | 0.000 | -0.001 |
| GLU-35 | 0.000 | -0.001 |
| MET-36 | 0.000 | -0.001 |
| SER-37 | 0.000 | -0.000 |
| LEU-38 | 0.000 | -0.001 |
| PRO-39 | 0.000 | -0.000 |
| GLY-40 | 0.000 | -0.000 |
| ARG-41 | 0.000 | -0.001 |
| TRP-42 | 0.000 | -0.001 |
| LYS-43 | 0.000 | -0.001 |
| PRO-44 | 0.000 | -0.000 |
| LYS-45 | 0.000 | -0.001 |
| MET-46 | 0.000 | -0.001 |
| ILE-47 | -0.301 | -0.225 |
| GLY-48 | -0.940 | -0.830 |
| GLY-49 | -0.421 | -0.366 |
| ILE-50 | -0.722 | -0.764 |
| GLY-51 | 0.000 | -0.000 |
| GLY-52 | 0.000 | -0.000 |
| PHE-53 | 0.000 | -0.001 |
| ILE-54 | 0.000 | -0.001 |
| LYS-55 | 0.000 | -0.001 |
| VAL-56 | 0.000 | -0.001 |
| ARG-57 | 0.000 | -0.001 |
| GLN-58 | 0.000 | -0.001 |
| TYR-59 | 0.000 | -0.001 |
| ASP-60 | 0.000 | -0.000 |
| GLN-61 | 0.000 | -0.001 |
| ILE-62 | 0.000 | -0.001 |
| LEU-63 | 0.000 | -0.001 |
| ILE-64 | 0.000 | -0.001 |
| GLU-65 | 0.000 | -0.001 |
| ILE-66 | 0.000 | -0.001 |
| CYS-67 | 0.000 | -0.000 |
| GLY-68 | 0.000 | -0.000 |
| HIS-69 | 0.000 | -0.001 |
| LYS-70 | 0.000 | -0.001 |
| ALA-71 | 0.000 | -0.000 |
| ILE-72 | 0.000 | -0.001 |
| GLY-73 | 0.000 | -0.000 |
| THR-74 | 0.000 | -0.000 |
| VAL-75 | 0.000 | -0.001 |
| LEU-76 | 0.000 | -0.001 |
| VAL-77 | 0.000 | -0.001 |
| GLY-78 | 0.000 | -0.000 |
| PRO-79 | 0.000 | -0.000 |
| THR-80 | 0.000 | -0.000 |
| PRO-81 | -0.120 | -0.129 |
| VAL-82 | -0.361 | -0.374 |
| ASN-83 | 0.000 | -0.000 |
| ILE-84 | -0.361 | -0.205 |
| ILE-85 | 0.000 | -0.001 |
| GLY-86 | 0.000 | -0.000 |
| ARG-87 | 0.000 | -0.001 |
| ASN-88 | 0.000 | -0.000 |
| LEU-89 | 0.000 | -0.001 |
| LEU-90 | 0.000 | -0.001 |
| THR-91 | 0.000 | -0.000 |
| GLN-92 | 0.000 | -0.001 |
| ILE-93 | 0.000 | -0.001 |
| GLY-94 | 0.000 | -0.000 |
| CYS-95 | 0.000 | -0.000 |
| THR-96 | 0.000 | -0.000 |
| LEU-97 | 0.000 | -0.001 |
| ASN-98 | 0.000 | -0.000 |
| PHE-99 | 0.000 | -0.001 |
| PRO-101 | 0.000 | -0.001 |
| GLN-102 | 0.000 | -0.001 |
| ILE-103 | 0.000 | -0.001 |
| THR-104 | 0.000 | -0.000 |
| LEU-105 | 0.000 | -0.001 |
| TRP-106 | 0.000 | -0.001 |
| GLN-107 | 0.000 | -0.001 |
| ARG-108 | -0.666 | -0.536 |
| PRO-109 | 0.000 | -0.000 |
| LEU-110 | 0.000 | -0.001 |
| VAL-111 | 0.000 | -0.001 |
| THR-112 | 0.000 | -0.000 |
| ILE-113 | 0.000 | -0.001 |
| LYS-114 | 0.000 | -0.001 |
| ILE-115 | 0.000 | -0.001 |
| GLY-116 | 0.000 | -0.000 |
| GLY-117 | 0.000 | -0.000 |
| GLN-118 | 0.000 | -0.001 |
| LEU-119 | 0.000 | -0.001 |
| LYS-120 | 0.000 | -0.001 |
| GLU-121 | 0.000 | -0.001 |
| ALA-122 | 0.000 | -0.000 |
| LEU-123 | -0.146 | -0.085 |
| LEU-124 | 0.000 | -0.001 |
| ASP-125 | -0.228 | -0.234 |
| THR-126 | 0.000 | -0.000 |
| GLY-127 | -0.607 | -0.623 |
| ALA-128 | -0.507 | -0.302 |
| ASP-129 | -0.703 | -0.562 |
| ASP-130 | -0.272 | -0.232 |
| THR-131 | 0.000 | -0.000 |
| VAL-132 | -0.120 | -0.138 |
| LEU-133 | 0.000 | -0.001 |
| GLU-134 | 0.000 | -0.001 |
| GLU-135 | 0.000 | -0.001 |
| MET-136 | 0.000 | -0.001 |
| SER-137 | 0.000 | -0.000 |
| LEU-138 | 0.000 | -0.001 |
| PRO-139 | 0.000 | -0.000 |
| GLY-140 | 0.000 | -0.000 |
| ARG-141 | 0.000 | -0.001 |
| TRP-142 | 0.000 | -0.001 |
| LYS-143 | 0.000 | -0.001 |
| PRO-144 | 0.000 | -0.000 |
| LYS-145 | 0.000 | -0.002 |
| MET-146 | 0.000 | -0.001 |
| ILE-147 | -0.421 | -0.501 |
| GLY-148 | -0.744 | -0.553 |
| GLY-149 | -0.326 | -0.260 |
| ILE-150 | -1.023 | -0.894 |
| GLY-151 | 0.000 | -0.000 |
| GLY-152 | 0.000 | -0.000 |
| PHE-153 | 0.000 | -0.001 |
| ILE-154 | 0.000 | -0.001 |
| LYS-155 | 0.000 | -0.001 |
| VAL-156 | 0.000 | -0.001 |
| ARG-157 | 0.000 | -0.001 |
| GLN-158 | 0.000 | -0.001 |
| TYR-159 | 0.000 | -0.001 |
| ASP-160 | 0.000 | -0.000 |
| GLN-161 | 0.000 | -0.001 |
| ILE-162 | 0.000 | -0.001 |
| LEU-163 | 0.000 | -0.001 |
| ILE-164 | 0.000 | -0.001 |
| GLU-165 | 0.000 | -0.001 |
| ILE-166 | 0.000 | -0.001 |
| CYS-167 | 0.000 | -0.000 |
| GLY-168 | 0.000 | -0.000 |
| HIS-169 | 0.000 | -0.001 |
| LYS-170 | 0.000 | -0.001 |
| ALA-171 | 0.000 | -0.000 |
| ILE-172 | 0.000 | -0.001 |
| GLY-173 | 0.000 | -0.000 |
| THR-174 | 0.000 | -0.000 |
| VAL-175 | 0.000 | -0.001 |
| LEU-176 | 0.000 | -0.019 |
| VAL-177 | 0.000 | -0.001 |
| GLY-178 | 0.000 | -0.000 |
| PRO-179 | 0.000 | -0.000 |
| THR-180 | 0.000 | -0.000 |
| PRO-181 | -0.326 | -0.304 |
| VAL-182 | -0.326 | -0.317 |
| ASN-183 | 0.000 | -0.000 |
| ILE-184 | -0.301 | -0.247 |
| ILE-185 | 0.000 | -0.001 |
| GLY-186 | 0.000 | -0.000 |
| ARG-187 | 0.000 | -0.001 |
| ASN-188 | 0.000 | -0.000 |
| LEU-189 | 0.000 | -0.001 |
| LEU-190 | 0.000 | -0.001 |
| THR-191 | 0.000 | -0.000 |
| GLN-192 | 0.000 | -0.001 |
| ILE-193 | 0.000 | -0.001 |
| GLY-194 | 0.000 | -0.000 |
| CYS-195 | 0.000 | -0.000 |
| THR-196 | 0.000 | -0.000 |
| LEU-197 | 0.000 | -0.001 |
| ASN-198 | 0.000 | -0.000 |
| PHE-199 | 0.000 | -0.001 |
| BEC-200 | -19.522 | -23.048 |
能量分解图示
收敛性测试
对于分解后的各个能量, 只有PB能量对选项的设置比较敏感, 使用默认参数得到的值未必是收敛的. 因此最好进行收敛性测试, 确定每个选项的最佳设置.
计算PB能量时, 有三个选项影响很大, cfac, fadd, df. 下面列出了这三个选项取不同值时, 所得的PB能量.
| df | fadd | cfac | dPB | PBcom | PBpro | PBlig | ||
|---|---|---|---|---|---|---|---|---|
| 0.125 | 10 | 3 | 286.890 | -5433.068 | -5583.091 | -136.867 | ||
| 0.2 | 10 | 3 | 289.313 | -5471.557 | -5622.779 | -138.091 | ||
| 0.25 | 10 | 3 | 292.329 | -5505.595 | -5658.472 | -139.451 | ||
| 0.5 | 10 | 3 | 330.673 | -5858.081 | -6030.070 | -158.685 | ||
| 0.6 | 10 | 3 | 337.288 | -6047.515 | -6223.224 | -161.579 | ||
| 0.7 | 10 | 3 | 358.489 | -6207.564 | -6393.435 | -172.618 | ||
| 0.8 | 10 | 3 | 328.954 | -6198.508 | -6366.897 | -160.565 | ||
| 0.9 | 10 | 3 | 328.954 | -6198.508 | -6366.897 | -160.565 | ||
| 1 | 10 | 3 | 328.954 | -6198.508 | -6366.897 | -160.565 | ||
| 0.125 | 10 | 2 | 286.890 | -5433.074 | -5583.097 | -136.867 | ||
| 0.125 | 5 | 1.5 | 286.807 | -5433.602 | -5583.554 | -136.856 | ||
| 0.125 | 5 | 2 | 286.808 | -5433.556 | -5583.508 | -136.856 | ||
| 0.125 | 5 | 3 | 286.808 | -5433.548 | -5583.500 | -136.856 | ||
| 0.2 | 5 | 1.5 | 289.638 | -5474.439 | -5625.939 | -138.138 | ||
| 基于每帧的网格设置 | ||||||||
| 0.2 | 10 | 3 | 289.526 | -5471.557 | -5622.779 | -138.304 | ||
| 0.5 | 10 | 3 | 333.012 | -5858.081 | -6030.070 | -161.023 | ||
| df 影响 | ||||||||
| 0.12 | 5 | 1.5 | 286.626 | -5429.042 | -5578.939 | -136.728 | ||
| 0.13 | 5 | 1.5 | 286.953 | -5434.188 | -5584.351 | -136.789 | ||
| 0.14 | 5 | 1.5 | 287.2 | -5440.064 | -5590.183 | -137.081 | ||
| 0.15 | 5 | 1.5 | 287.629 | -5444.356 | -5594.904 | -137.081 | ||
| 0.2 | 5 | 1.5 | 290.160 | -5474.426 | -5626.27 | -138.316 | ||
| 0.25 | 5 | 1.5 | 293.828 | -5500.399 | -5653.894 | -140.332 | ||
| 0.3 | 5 | 1.5 | 295.971 | -5549.837 | -5704.235 | -141.573 | ||
| 0.35 | 5 | 1.5 | 300.274 | -5613.973 | -5772.673 | -141.573 | ||
| 0.5 | 5 | 1.5 | 326.286 | -5758.149 | -5923.278 | -161.158 | ||
| fadd 影响 | ||||||||
| 2 | 5 | 1.5 | 290.160 | -5474.426 | -5626.27 | -138.316 | ||
| 2 | 10 | 1.5 | 290.076 | -5470.877 | -5622.549 | -138.403 | ||
| 2 | 15 | 1.5 | 289.859 | -5467.796 | -5619.252 | -138.403 | ||
| 2 | 20 | 1.5 | 290.447 | -5473.721 | -5625.765 | -138.403 | ||
| 2 | 25 | 1.5 | 289.960 | -5472.684 | -5624.241 | -138.403 | ||
| cfac 影响 | ||||||||
| 2 | 5 | 1.5 | 290.160 | -5474.426 | -5626.27 | -138.316 | ||
| 2 | 5 | 2 | 290.150 | -5474.377 | -5626.221 | -138.306 | ||
| 2 | 5 | 2.5 | 290.149 | -5474.358 | -5626.202 | -138.304 | ||
| 2 | 5 | 3 | 290.148 | -5474.384 | -5626.229 | -138.304 | ||
可见, 网格大小和格点间距对最终结果的影响很大. 对此, 引用一下我咨询专家时的答复.
邮件1
有一条分子动力学模拟得到的轨迹, 是蛋白和配体的, 含有很多帧. 如果计算蛋白和配体之间的PB作用能, 我需要分别计算蛋白+配体, 蛋白, 配体三个体系的PB能量, 然后做差值获得PB相互作用能. 在计算每个体系的时候都要设置网格大小, 而且使用APBS的话, 还需要设置两套网格, 粗糙网格和细密网格.
我的第一个问题是, 只算一帧构型的PB相互作用能的话, 对这三个体系是不是 必须 使用相同的网格大小和格点间距, 还是可以对每个体系使用独立的, 只与其自身坐标有关的网格大小和格点间距? 不考虑计算效率的话, 哪种方法更正确?
如果前一个问题的答案是, 对每帧的三个体系必须使用相同网格大小和格点间距, 那我下面的问题是, 如果我要算很多帧构型的PB相互作用能, 那所有这些帧是不是也要使用相同的网格大小和格点间距, 也就是说我们需要对整条轨迹使用相同的网格大小和格点间距? 是否这样得到的结果更自洽?
我没有很细致的检验过APBS的细节,不过我觉得/感觉,如果 网格取得充分大(粗网格能cover两倍的分子大小以上,对于溶剂化能的计算细网格能cover整个分子那我认为应该是够了 —- 而有的只关心反应活性部位的电场计算,那细网格只要包含比活性部位大一点就行了),格点间距取得足够小(比如0.2 A 应该够了),并且如果你每次算的是 分子的 solvation energy (这样它内部已经解了两次 方程),那么我觉的每次计算(三个体系)应该不需要使用同样的网格,就是说可以各自独立的采用粗网格和细网格,因为这时我们“认为”单个体系APBS计算是准确的。 你可以计算试试看。 这样的话,那么对一帧的计算还是整条轨迹多帧的计算应该都没关系,可独立计算。不过,感觉上对多帧的同一个体系比如复合物,最好采用一样的网格精度,以减少网格精度上到带来互相间的计算差别。
当然,对于一帧中的三个体系的计算,我觉得,如果你每次计算都采用同一套网格来计算,并且网格大小满足 我上面讲的覆盖关系,那么这样当然会略为减少网格选取的不同(坐标系平移,转动,网格尺寸大小)带来数值误差,我估计这个偏差较小(如果像上面说的计算正确的话),但这样就是计算量大(因为你计算单体和复合体时用的是一样大的网格系统)。
这些建议你要设计实验来验证一下
邮件2
选择了一个含200个氨基酸残基的蛋白, 含90个原子的配体的体系进行了一些测试, 结果如下:
(见上表)
大致可以看到, 影响最大的是格点间距, 其他粗细网格的增加值影响小, 如您所言, 只要能覆盖即可. 此外, 使用整体一致的网格还是单独的网格对这个体系影响也很小,.
对太大的蛋白, 我们没法使用很小的网格, 所以我检验了一下, 在网格大小固定, 格点间距不太大的情况下, PB相互作用能大致与格点间距的三次方成正比. 我想问下, 这个关系是否正确, 还是理论上有其他更正确的关系式? 如果有的话, 这种关系式对格点间距适用范围的最大值如何确定? 如果这些问题都能解决的话, 我们就可以做外推得到格点间距为零时的PB相互作用能, 这样就可以避免格点间距设置的影响了.
你前面的观察应该合理的。
后半部分你说 “PB相互作用能大致与格点间距的三次方成正比”,这个应该不对,因为如果这样,格点为零,则PB能为0,当然不是。可能的话也是“PB相互作用能的误差大致与格点间距的三次方成正比”,但这个我的印象里也没有看到有人这么说(当然我关于APBS的文章读的很少)。一般来说是这样,一个软件的计算误差由它的算法决定,对于普通线性有限元来说,它的“解的误差”(L2 norm)与网格尺寸(大致可认为类似于格点间距)成平方关系(二阶),有限差分方法或有限体方法的误差也与具体算法格式(方法)有关,一般的方法对一般光滑问题的解的误差也能达到二阶精度,达到三阶需要更精细一点的方法,但有些问题的有限差分方法的解随着格点间距的减小并不能总是保证一致的精度阶,有时候随着格点间距减小误差甚至会变得更大。 你用的APBS的方法应该是用其中的有限差分方法,其实可能应该说有限体方法更恰当。APBS的计算里面还有奇异电荷分配的问题,还有粗细网格同时用的问题,所以具体的误差阶我没有研究过,不知道它是几阶,大致的三阶也是有可能的。不过就算是这样,你要用这个关系也挺麻烦,因为每次计算你想得到精确值就要多计算若干次得到准确的三阶估计才能用。
源文件链接:
https://jerkwin.github.io/2019/07/31/gmx_mmpbsa%E4%BD%BF%E7%94%A8%E8%AF%B4%E6%98%8E/